- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Mécanismes de développement déclenchés par les ARNnc
Équipe REGARN / Martin Crespi
Dans ce deuxième thème, nous avons cherché à mieux comprendre les mécanismes par lesquels les ARNnc peuvent affecter le développement et la base moléculaire de l’interaction des ARNnc avec les ribonucléoprotéines spécifiques afin de modifier l’expression génétique cible. Dans le cadre du projet EPIRNA labex SPS (coordinateur M. Crespi), nous avons identifié le long ARNnc ASCO (pour « Alternative Splicing Competitor ») qui peu interagir avec les régulateurs nucléaires de l'épissage AtNSRa et AtNSRb (pour Nuclear Speckle RNA-binding protein). Il est intéressant de noter qu’en modulant l’expression de ce long ARNnc, nous avons modifié les motifs d’épissage alternatifs dépendant de NSR et la formation de racines latérales. Nous avons donc proposé que la modulation de l’épissage alternatif pourrait être un nouveau mécanisme de régulation médié par les longs ARNnc. Ces longs ARNnc « imiteraient » les introns et pourraient servir à moduler l’épissage alternatif pendant le développement (Bardou et al., 2014 ; Van Du et al., 2015). Il s’agit d’un mécanisme émergent chez les eucaryotes et il y a de plus en plus de preuves montrant que les ARNnc peuvent interférer avec la régulation de l’épissage alternatif (Romero-Barrios et al., 2018).
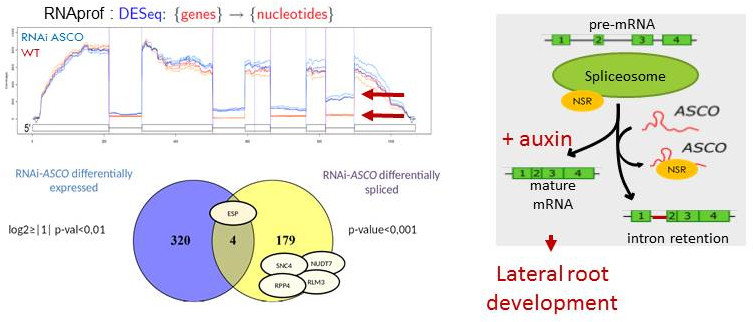
En plus de ces longs ARNnc « liés à l’épissage », nous avons identifié un autre groupe de long ARNnc exclusivement co-localisés avec des siARN de 24nt, connus pour être liés aux changements épigénétiques et à l’extinction des gènes. Il est intéressant de noter que l’extinction de l’un de ces longs ARNnc a entraîné la répression de son gène voisin PID (pour PINOID kinase), un régulateur majeur des réponses auxines dans les racines. Ensuite, nous avons montré que ce long ARNnc, appelé APOLO (pour Auxin PrOmoter-dependent LOop lncRNA), est capable de réguler une « boucle chromatinienne » allant de sa position jusqu’au gène cible afin de contrôler les états épigénétiques du promoteur cible de PID. En analysant les changements d’histones (en collaboration avec le groupe de Moussa Benhamed, IPS2), ainsi que l’état de la méthylation de l’ADN et la formation de la boucle chromatinienne en réponse à l'auxine, nous avons découvert qu’APOLO pourrait organiser la structure chromatinienne du promoteur de PID (Ariel et al., 2014). Ces travaux soutiennent l’idée que les long ARNnc nucléaires sont des acteurs clés dans la régulation de la structure 3D et des états épigénétiques de la chromatine, ouvrant des perspectives intéressantes tant pour les plantes que pour les animaux. Nous croyons que cette classe d’ARN non codants pourrait être un nouvel élément impliqué dans la topologie du génome et la structure de la chromatine (Ariel et al., 2015).
Nous avons également cherché des changements dans la reprogrammation épigénétique médiée par les ARNnc chez Medicago truncatula. La compréhension de la différenciation coordonnée des cellules végétales et bactériennes au cours de l’interaction symbiotique avec Sinorhizobium meliloti est une transition développementale accompagnée d’une reprogrammation massive de l’expression des gènes qui peut impliquer une régulation épigénétique. Comme nous l’avons mentionné, certains changements épigénétiques sont associés à la production de siARN de 24nt et de long ARNnc de type APOLO (Ariel et al., 2014). Dans nos travaux précédents (Formey et al., 2014), nous étions perplexes quant au fait que de nombreux siARN étaient spécifiquement exprimés pendant la nodulation. En collaboration avec le laboratoire de Pascal Gamas et J. Gouzy (LIPM Toulouse) dans le cadre du projet ANR EPISYM (coordinateur M. Crespi), nous avons identifié que, contrairement à MtDCL3, la principale enzyme impliquée dans la biogenèse des siARN de 24nt, une autre RNase III (appelée RTL1 pour RNAse-Three-L-like 1) est fortement régulée dans la zone de différenciation des nodules. Ces enzymes sont probablement capables de modifier in vivo de manière antagoniste le répertoire des siARN de 24nt dans les nodules de M. truncatula. Des analyses fonctionnelles les gènes DCL3 et RTL1 de Medicago suggèrent une association avec des phénotypes symbiotiques intéressants. En particulier, la répression de RTL1 par l’ARNi a fortement entravé le développement des nodules, leur capacité à fixer l’azote et leur viabilité bactérienne.
En raison de leur rôle dans le réglage fin de l’expression d’autres gènes, nous nous attendons à ce que les ARNnc modifient quantitativement le phénotype observé, soit en intensité, mais également au cours du temps. Pour nous aider, nous construisons actuellement un robot de phénotypage en collaboration avec le FabLab Digiteo. Il nous permettra de surveiller un plus grand nombre de plantes à une résolution temporelle plus élevée et donc de capturer les modifications fines du phénotype racinaire attendues lors de la modification de l’expression des ARNnc.
