- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Voies de signalisation du stress
Équipe STRESS / Jean Colcombet et Hannes Kollist
Les plantes sont fixées au sol et doivent composer avec les perturbations locales de leur environnement, c’est-à-dire les stress biotiques et abiotiques. Comprendre la manière dont elles perçoivent ces stress et s’y adaptent est un sujet de recherche majeur pour permettre l’élaboration des plantes d’intérêt agronomique du futur.
Une fois perçus, ces signaux activent des voies de signalisation intracellulaire qui font souvent intervenir des protéines kinases, enzymes capables de phosphoryler d’autres protéines pour en changer les propriétés. C’est un moyen très efficace et répandu pour transmettre une information à d’autres acteurs de la signalisation ou pour réguler des protéines impliquées dans la spécificité de réponse. Le génome des plantes code généralement pour au moins un millier de protéines kinases ce qui souligne bien l’importance de ce type de signalisation chez les plantes. Ces nombreuses kinases ont des caractéristiques variées et l’équipe « Signalisation du stress » a focalisé son travail sur deux grandes familles de kinases pour en comprendre les fonctions dans la signalisation et les réponses aux stress:
- les modules MAPKs composés d’une MAP3K, une MAP2K et une MAPK s’activent en série par phosphorylation. Chez les plantes les modules MAPKs sont codés par de grandes familles multi-géniques qui peuvent, en théorie, définir de nombreux modules fonctionnels de signalisation possibles. La plupart des études publiées se concentrent sur trois MAPKs, MPK3, MPK4 et MPK6, ainsi que leurs MAP2Ks et MAP3Ks correspondantes. Ces trois MAPKs s’activent rapidement en réponse à des stress à la fois biotiques et abiotiques et régulent différents mécanismes de défense comme la synthèse d’hormones et de phytoalexines ou la reprogrammation de l’expression génique. De plus ces mêmes kinases sont aussi impliquées dans différents aspects du développement végétal comme la formation des stomates, la cytocinèse et l’abscission. En comparaison peu de choses sont connues sur les autres membres de la famille MAPK.
- Les CDPKs qui comptent 34 membres chez Arabidopsis sont capables de traduire des élévations de concentrations calciques dans le noyau ou le cytoplasme en signaux de phosphorylation. Notre groupe travaille plus spécifiquement sur deux CDPKs, CPK5 et CPK6, qui sont deux régulateurs clefs des réponses aux stress. CPK5 et CPK6 permettent notamment aux plantes de résister à certaines maladies en régulant de manière redondante le burst oxydatif, la synthèse de phytoalexines et d’hormones, et les réponses transcriptionnelles. Mais les mécanismes moléculaires demeurent encore mal compris.
Les 6 axes de recherche du laboratoire
Le projet de recherche de l’équipe évolue constamment en fonction des données de la littérature, des résultats de l’équipe et de envies de chacun. Voilà quelques questions scientifiques auxquelles nous nous intéressons en ce moment :
Quels sont les rôles des CDPKs dans l’adaptation des plantes aux stress abiotiques ?
Marie Boudsocq
Les stress osmotiques générés par la sécheresse, la salinité des sols ou le froid sont des contraintes environnementales majeures qui affectent le développement des plantes et les rendements agricoles. Ils induisent une déshydratation au niveau cellulaire qui s’accompagne d’un déséquilibre ionique et d’un stress oxydatif. Pour maintenir le bon fonctionnement cellulaire, la plante active des voies de signalisation qui régulent l’expression génique, le métabolisme et les systèmes anti-oxydants. Le calcium est un second messager essentiel de ces réponses, qui est notamment décodé par les CDPKs. Nous avons montré que le double mutant cpk5,cpk6 était hypersensible au stress salin. Nous développons des approches omiques pour identifier les processus moléculaires mis en jeu.
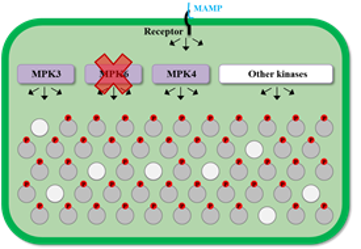
Quelle est l’implication des modules MAPK dans la régulation des réponses PTI et ETI ?
Julien Lang
Le module MPK3/6 est connu pour être activé de manière rapide et transitoire dans le cadre des réponses PTI ("PAMP-Triggered Immunity") et de manière retardée et durable dans le cadre des réponses ETI ("Effector-Triggered Immunity"). Nous cherchons ainsi à comprendre comment ces cinétiques d’activation différentes impactent les réponses de défense, notamment au niveau de la reprogrammation génique.
En outre nous avons identifié un nouveau module MAPK qui s’active de manière durable dans le cadre des réponses PTI. L’objectif est désormais de caractériser le rôle de ce nouveau module dans les réponses immunitaires et de déterminer son lien éventuel avec le module classique MPK3/6.
Quels sont les substrats des protéines kinases des modules de signalisation activés par les stress ?
Marie Boudsocq, Julien Lang, Jean Bigeard
Identifier les substrats des protéines kinases est essentiel pour déchiffrer les mécanismes moléculaires qui sous-tendent leurs fonctions biologiques. Pour cela, nous avons mis en œuvre deux stratégies complémentaires. D’une part, nous recherchons des interactants des protéines kinases, soit par identification de complexes in planta (TAP-MS), soit par crible en double-hybride de levure. D’autre part, nous menons des approches phosphoprotéomiques en comparant des génotypes sauvage/mutants ou en surexprimant de façon inductible des formes constitutivement actives des kinases. Les différents candidats obtenus sont ensuite validés avant de lancer une caractérisation fonctionnelle plus approfondie. Actuellement nous nous intéressons plus particulièrement à trois candidats : un facteur de remodelage de la chromatine impliqué dans les réponses immunitaires médiées par les voies MAPKs, ainsi qu’un inhibiteur de phosphatase et un facteur de transcription qui sont a priori des substrats communs aux voies MAPKs et CDPKs et qui offrent ainsi une opportunité nouvelle d’étudier les connexions entre ces deux voies de signalisation.
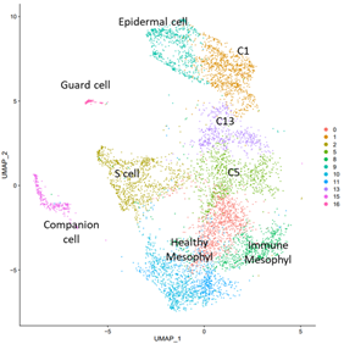
Peut-on décrire l’organisation des réponses immunitaires à l’échelle cellulaire à l’aide des technologies single cells ?
Julien Lang
Les réponses de défense des plantes mettent en jeu différents processus biologiques qui permettent à la plante de se défendre contre les attaques de pathogènes. La manière dont ces différents processus sont orchestrés au sein des organes végétaux et pourraient dépendre de types cellulaires particuliers est encore très mal comprise. Sur la base d’analyses single-cell RNAseq de feuilles d’Arabidopsis infectées par Pseudomonas syringae, nous cherchons à mieux caractériser les réponses immunitaires dépendantes de différents types cellulaires (cellules du mésophylle, de l’épiderme, de garde, et vasculaires), et à comprendre comment ces types cellulaires distincts peuvent converger vers des réponses de défense communes ou alternativement diverger entre différentes sortes de réponses. Nous analysons également comment un mutant des voies de signalisations MAPK, connu pour être immuno-déficient, se comporte au niveau single-cell en réponse au pathogène. Dans leur ensemble ces données proposent un cadre innovant pour décrire et explorer les réponses cellulaires à un stress biotique.
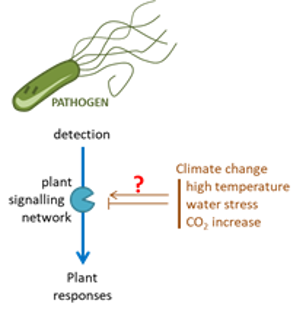
Quels seront les impacts du changement climatique sur la perception des stress biotiques par la plante ?
Jean Colcombet, Erwan Le Deunff
Les changements climatiques en cours sont susceptibles de perturber la sécurité alimentaire mondiale et déstabiliser les sociétés. En plus de canicules et sécheresses plus nombreuses, l’augmentation des températures provoque la migration vers les pôles de pathogènes des cultures autrefois tropicaux. Les pertes de rendement liées aux stress biotiques, déjà importantes actuellement, risquent d’encore croitre à l’avenir et seront par ailleurs exacerbées par la sécheresse et les hautes températures. Or si la signalisation et les réponses des plantes à l’interaction avec des pathogènes sont assez bien décrites aujourd’hui, l’effet du changement climatique et notamment de l’augmentation des températures sur ces processus reste très peu documenté. Le laboratoire s’est récemment rendu compte que certains des principaux modules du réseau de signalisation activé par les pathogènes étaient affectés par les hautes températures. Notre hypothèse est que les réponses aux pathogènes en seront largement reprogrammées. Notre travail vise à mieux appréhender les conséquences de la température sur l’aptitude des plantes à résister aux stress biotiques.
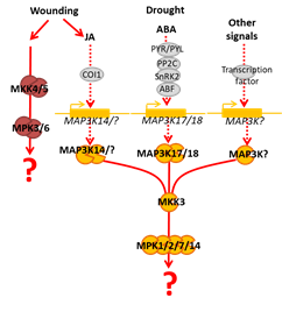
Quel est le rôle du module MKK3 dans l’intégration des contraintes environnementales ?
Jean Colcombet
Bien que les familles MAP3K, MAP2K et MAPK incluent de nombreux membres, peu de modules MAPK complets ont été caractérisés jusqu’à présent. En 2015 nous avons publié la découverte d’un nouveau module MAPK complet constitué de MAP3K17/18-MKK3-MPK1/2/7/14, module activé par l’acide abscissique (ABA) et jouant un rôle dans l’adaptation de la plante à des conditions de sécheresse (Danquah et al. 2015 Plant J, de Zelicourt et al. 2016 TIPS). Nous avons également montré que contrairement à d’autres MAPKs qui sont généralement activées en quelques minutes après exposition au stress, MPK1/2/7/14 sont activées par l’ABA après plusieurs heures. Cette activation dépend de la synthèse de novo de MAP3K17/18 dont les niveaux d’expression demeurent indétectables en absence de stress (Danquah et al. 2015 Plant J, Boudsocq et al.2015 PSB). A notre connaissance il s’agit de la première description d’un tel module MAPK aussi bien chez les végétaux que chez les animaux (Colcombet et al. 2016 Front. In Plant Science).
Mais il semble que ce type de modules ait une fonction plus générale. Le laboratoire a en effet montré que la blessure, un stress très courant pour les plantes, était elle aussi capable de l’activer (Sözen et al. 2020, Plant Cell). Dans ce cas-là, c’est principalement le gène MAP3K14 qui est fortement up-régulé et responsable de l’activation des kinases en aval. Ce travail a aussi été l’occasion de montrer que l’activation lente du module MKK3 était indépendante de celle, rapide, du module MKK4/5-MPK3/6, connue depuis 20 ans. Dans le cadre d’une collaboration avec Axel Mithöfer, nous avons enfin montré que des chenilles du papillon Spodoptera littoralis se développaient mieux sur des plantes mutées pour le module MKK3, indiquant un rôle important de résistance aux insectes phytophages.
Ce projet de recherche se poursuit avec la découverte et caractérisation des nombreux contextes environnementaux dans lesquels le module MKK3 est impliqué. Nous pensons aujourd’hui qu’il a un rôle beaucoup plus général que l’on imaginait à l’origine. Ceci est notamment étayé par le constat que des gènes codant pour les kinases des modules MKK3 sont retrouvés systématiquement au sein de tous les génomes végétaux, des bryophytes aux angiospermes, et que les MAP3K de la famille de MAP3K14/17/18 sont fortement régulés par de nombreux stress (Colcombet et al. 2016 Front. In Plant Science). Toutefois, la question de savoir ce qu’il contrôle dans la cellule reste à ce jour sans réponse…