- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Expression génomique des organites
Notre objectif principal est de comprendre la régulation de l'expression des gènes dans les organites
La compréhension des fonctions complexes et des mécanismes de régulation au sein des organites des plantes est fondamentale pour améliorer la productivité des cultures et renforcer leur résilience aux conditions environnementales variables. Les plastes et les mitochondries, principaux sites du métabolisme énergétique dans les cellules eucaryotes, exercent également une influence considérable sur diverses voies métaboliques. Au cours de l'évolution, ces organites ont subi une réduction substantielle de leur génome après les événements d’endosymbiose, ce qui a conduit au transfert du matériel génétique vers le noyau de la cellule. Par conséquent, les processus métaboliques au sein des organites dépendent fortement des protéines codées par le noyau et ciblées par la suite sur ces organites. Néanmoins, ces organites ont conservé des génomes compacts qui abritent des protéines et des ARN essentiels à leurs fonctions biologiques, ce qui nécessite un contrôle coordonné de l'expression des gènes nucléaires et des organites. Divers processus régissant les activités transcriptionnelles et post-transcriptionnelles des organites, notamment la réplication de l'ADN, la transcription de l'ARN, le traitement de l'ARN, l'édition de l'ARN, l'épissage de l'ARN et la traduction, reposent sur des protéines codées dans le noyau de la cellule. Parmi ces facteurs nucléaires, la grande famille des protéines à domaines pentatricopeptides répétés (PPR) constitue un élément central dans l'orchestration de la régulation des génomes des organites. Ces protéines codées dans le noyau sont presque exclusivement dirigées vers les mitochondries ou les chloroplastes, où elles se lient spécifiquement aux molécules d'ARN, participant activement à toutes les étapes de l'expression des gènes, de la transcription à la traduction. En complément des PPR, des ribonucléases, à la fois endo- et exo-ribonucléases, contribuent à la mautration des transcripts naissants, aboutissant à la formation du transcriptome mature des organites. Ce réseau complexe d'interactions (protéine-ARN, protéine-protéine, ADN-protéine) est dynamique et se déroule principalement dans le nucléoïde, un complexe nucléo-protéique encore mal caractérisé.
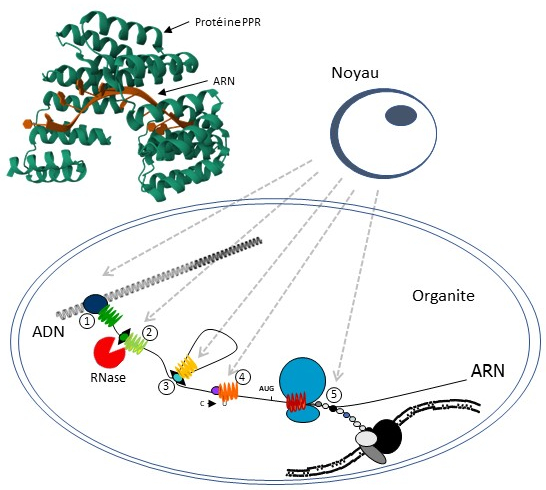
Historiquement, notre équipe a joué un rôle pionnier dans la découverte de la famille PPR en 2000. Par la suite, nous avons effectué des caractérisations génomiques de cette famille chez Arabidopsis, le riz et le Physcomitrium. Nous avons également entrepris diverses études génétiques et fonctionnelles sur les membres de la famille Arabidopsis. Au fil du temps, nos recherches se sont élargies aux ribonucléases plastidiales et à l'étude de l'expression des gènes des organites dans des contextes dynamiques, par exemple au cours du développement ou dans des conditions de stress, dans le cadre de trois projets principaux :
- Acteurs de l’expression génétique des organites
- Organites au cœur de la réponse des plantes aux stress
- Organites et différentiation C4
En parallèle, l'équipe OGE développe des outils de génomique fonctionnelle disponibles pour la communauté végétale à travers l'activité de deux plateformes technologiques : la Plateforme Transcriptomique Plantes Paris-Saclay et la Plateforme SPOmics Interactome.
La plateforme SPOmics Interactome est née de la participation active de l'équipe OGE à un projet international de cartographie de l'interactome d'Arabidopsis. Cet effort de collaboration a été coordonné par les professeurs Joseph Ecker (Salk Institute, San Diego) et Marc Vidal (DFCI, Boston). La carte du réseau d'interactions des protéines végétales a été construite à l'aide d'un test Y2H amélioré, développé à l'origine au DFCI pour l'étude des interactions protéiques dans les protéines de C. elegans, de l'homme et de la levure. L'ensemble de données initiales de ce projet a été publié en 2011 (AIM, 2011). Il présentait une carte complète des interactions binaires protéine-protéine, englobant environ 6200 interactions de haute qualité entre environ 2700 protéines.
