- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Projet « réseau d’interaction protéine-protéine du chloroplaste »
La plateforme InterATOME (IPS2) a été récemment impliquée dans le projet international « Arabidopsis Interactome Mapping – AIM » pour établir la première carte du réseau d'interactions protéiques des plantes, ainsi que dans la construction de la plus grande base de données d'interacteurs d'effecteurs de pathogènes au travers de plusieurs collaborations internationales.
Chez les eucaryotes, l'origine endosymbiotique des cellules et la séparation inhérente des compartiments intracellulaires nécessitent une communication efficace entre les organites et le noyau. De nombreuses données suggèrent que le réticulum endoplasmique (RE) et les chloroplastes sont biochimiquement connectés, ce qui permet, entre autres, l'échange de peroxyde d'hydrogène (H2O2). Ce composé peut également induire la formation de stromules plastidiaux (protubérances tubulaires remplies de stroma s'étendant à partir du corps principal du chloroplaste) pour transmettre des signaux au noyau. Ces signaux conduisent à l'activation de voies rétrogrades et, si la production de ROS est suffisamment élevée, à la mort cellulaire.
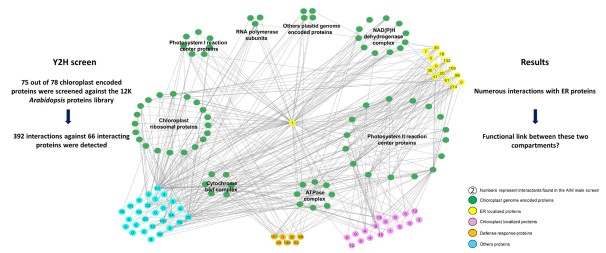
La plateforme InterATOME a récemment synthétisé l'ensemble des gènes codants du chloroplaste d'Arabidopsis afin de générer le premier réseau d'interaction protéine-protéine du chloroplaste à l'aide d'un système de double hybride sur levures à large échelle. Les résultats préliminaires, correspondant au crible de 75 des 78 protéines codées par le génome du chloroplaste contre la collection de 12 000 protéines d'Arabidopsis codées par le noyau, révèlent plusieurs interactions entre les protéines du chloroplaste et du RE impliquées dans la réponse aux stress biotiques et abiotiques et dans les voies de signalisation rétrograde. Ces résultats soulignent l'existence de liens étroits entre les chloroplastes et le RE.
25/09/2023
