- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
MUTAGENESES
TILLING-NGS
Le TILLING (Targeting Induced Local Lesions in Genomes) est une approche de génétique inverse, développée sur la plateforme EPITRANS comme outil de validation fonctionnelle chez des espèces cultivées pour la plupart récalcitrantes à la transformation génétique. Cette technologie permet à nos partenaires de valider la fonction d’un gène directement sur l’espèce cultivée travaillée et d’identifier de nouveaux allèles d’intérêts agronomiques, non OGM.
La plateforme EPITRANS produit et exploite des collections de mutants induites à l’EMS qui constituent aujourd’hui une ressource de plus de 250.000 familles sur une dizaine d’espèces cultivées (melon, concombre, pastèque, courgette, pois, sauge, papaye, lin, poivron et tomate) et une espèce modèle (Brachypodium) et développe également des systèmes de détections de mutations avec les technologies les plus innovantes (séquençage haut-débit, PCR digitale, etc).
Le système de détection de mutations utilisé en TILLING est basé sur le séquençage NGS de librairies de fragments PCR, étiquetés par des séquences uniques et amplifiés à partir d’échantillons poolés en plusieurs dimensions. Les lignées mutantes sont ensuite identifiées en utilisant le logiciel SENTINEL développé par la plateforme et protégé par l’INRAE.
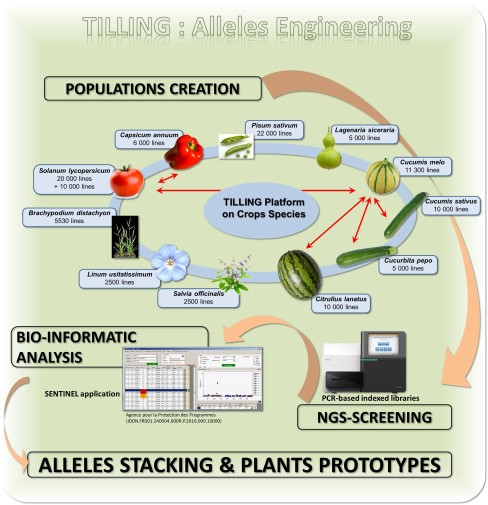
Le savoir-faire et les ressources TILLING multi-espèces proposés par la plate-forme EPITRANS sont uniques aux niveaux européen et international.
Pour cette application, la plateforme offre 3 niveaux de savoir-faire:
- La détection de mutation dans des régions génomiques ciblées déterminées par le partenaire. Le choix de la collection de mutants à cribler est discuté préalablement avec le partenaire en fonction de ses besoins.
- Le 2ème niveau est le même que le précèdent à la différence que le partenaire fourni les ADNs de la collection de mutants à cribler.
- Le 3ème niveau consiste à générer des mutants pour un caractère agronomique donné. La plateforme réalise alors la recherche de gènes candidats, la détection de mutations et la validation fonctionnelle. Ce type de contrat est réservé aux compagnies privées.
A ce jour, plus d’une quarantaine d’articles a été publié grâce à cette approche et 4 brevets ont été déposés.
Articles Ressources:
- A cucurbit androecy gene reveals how unisexual flowers develop and dioecy emerges. Boualem A, Troadec C, Camps C, Lemhemdi A, Morin H, Sari MA, Fraenkel-Zagouri R, Kovalski I, Dogimont C, Perl-Treves R, Bendahmane A. Science. 2015 Nov 6;350(6261):688-91.
- First TILLING platform in Cucurbita pepo: a new mutant resource for gene function and crop improvement. Vicente-Dólera N, Troadec C, Moya M, del Río-Celestino M, Pomares-Viciana T, Bendahmane A, Picó B, Román B, Gómez P. PLoS One. 2014. 9(11):e112743
- Development of a Cucumis sativus TILLinG platform for forward and reverse genetics. Boualem A, Fleurier S, Troadec C, Audigier P, Kumar AP, Chatterjee M, Alsadon AA, Sadder MT, Wahb-Allah MA, Al-Doss AA, Bendahmane A. PLoS One. 2014. 9(5):e97963
- PT-Flax (phenotyping and TILLinG of flax): development of a flax (Linum usitatissimum L.) mutant population and TILLinG platform for forward and reverse genetics. Chantreau M, Grec S, Gutierrez L, Dalmais M, Pineau C, Demailly H, Paysant-Leroux C, Tavernier R, Trouvé JP, Chatterjee M, Guillot X, Brunaud V, Chabbert B, van Wuytswinkel O, Bendahmane A, Thomasset B, Hawkins S. BMC Plant Biol. 2013 Oct 15;13:159.
- A TILLING platform for functional genomics in Brachypodium distachyon. Dalmais M, Darracq O, Ho-Yu-Kuang S, Wang Y, Bouvier d’Yvoire M, Antelme S, Cézard L, Legée F, Blondet E, Oria N, Troadec C, Brunaud V, Jouanin L, Höfte H, Bendahmane A, Lapierre C and Sibout R. PLoS One. 2013 Jun 19;8(6):e65503
- Engineering melon plants with improved fruit shelf life using the TILLING approach. Dahmani-Mardas F, Troadec C, Boualem A, Lévêque S, Alsadon AA, Aldoss AA, Dogimont C, Bendahmane A. PLoS One 2010 Dec 30;5(12)
- An induced mutation in tomato eIF4E leads to immunity to two potyviruses. Piron F, Nicolaï M, Minoïa S, Piednoir E, Moretti A, Salgues A, Zamir D, Caranta C, Bendahmane A. PLoS One. 2010 Jun 25;5(6):e11313.
- A new mutant genetic resource for tomato crop improvement by TILLING technology. Minoia S, Petrozza A, D'Onofrio O, Piron F, Mosca G, Sozio G, Cellini F, Bendahmane A, Carriero F. BMC Res Notes. 2010 Mar 12;3:69.
- A transposon-induced epigenetic change leads to sex determination in melon. Martin A, Troadec C, Boualem A, Rajab M, Fernandez R, Morin H, Pitrat M, Dogimont C, Bendahmane A. Nature. 2009 Oct 22;461(7267):1135-8.
- UTILLdb, a Pisum sativum in silico forward and reverse genetics tool. Dalmais M, Schmidt J, Le Signor C, Moussy F, Burstin J, Savois V, Aubert G, Brunaud V, de Oliveira Y, Guichard C, Thompson R, Bendahmane A. Genome Biol. 2008;9(2):R43.
- A conserved mutation in an ethylene biosynthesis enzyme leads to andromonoecy in melons. Boualem A, Fergany M, Fernandez R, Troadec C, Martin A, Morin H, Sari MA, Collin F, Flowers JM, Pitrat M, Purugganan MD, Dogimont C, Bendahmane A. Science. 2008 Aug 8;321(5890):836-8.
- Mutation detection using ENDO1: application to disease diagnostics in humans and TILLING and Eco-TILLING in plants. Triques K, Piednoir E, Dalmais M, Schmidt J, Le Signor C, Sharkey M, Caboche M, Sturbois B, Bendahmane A. BMC Mol Biol. 2008 Apr 23;9:42
- Characterization of Arabidopsis thaliana mismatch specific endonucleases: application to mutation discovery by TILLING in pea. Triques K, Sturbois B, Gallais S, Dalmais M, Chauvin S, Clepet C, Aubourg S, Rameau C, Caboche M, Bendahmane A. Plant J. 2007 Sep;51(6):1116-25
Brevets:
- Method for producing highly sensitive endonucleases, novel preparations of endonucleases and uses thereof. Bendahmane A, Triques K., Sturbois B., Aubourg S., Caboche M., (2004) PCT/EP2005/009220
- Genetic system for controlling the floral development of a dicotyledonous plant, and use thereof in detection and selection processes. Bendahmane A, Fergany M, Boualem A, Dogimont C(2007) PCT/FR2007/051197
- Combination of 2 genetic elements for controlling the floral development of a dicotyledonous plant, and use in detection and selection processes. Bendahmane A, Martin A, Troadec C, Dogimont C (2008) .PCT/FR2009/051510
MUTAGENESE SATURANTE
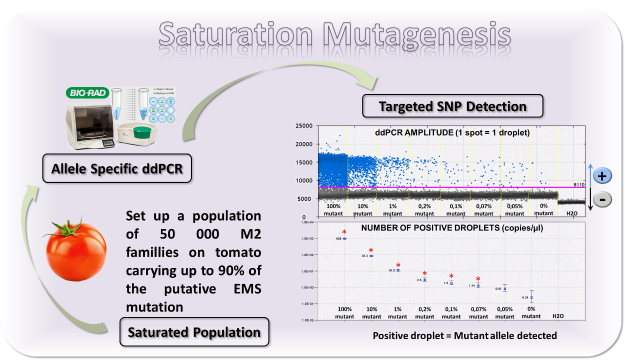
La mutagenèse aléatoire induite par des agents chimiques comme l’EMS est une approche simple et efficace pour saturer un génome entier en mutations et par conséquent, se rapprocher d’une approche de mutagenèse ciblée, encore inexistante chez les plantes cultivées récalcitrantes à la transformation génétique. La dose d’agent mutagène ne pouvant être augmentée sans affecter la santé et la reproduction de la plante, l’objectif est de produire des collections suffisamment grandes pour atteindre une probabilité supérieure à 90% de trouver une mutation à chaque G ou C du génome (transition nucléotidique générée par l’EMS).
Un projet pilote a été initié pour produire une collection de mutants saturantes chez la tomate (projet SAMUTAGENE) et pour développer un système de détection de mutants à très haut débit et très sensible, basé sur la technologie de PCR digitale en gouttelettes. Pour ce projet, partant de l’expertise de la plateforme EPITRANS en TILLING, l’objectif est de produire 50 000 familles mutées pour détecter probablement plus de 94% des G et C mutés dans le génome.
Lorsque le système de détection sera validé, il pourra être élargi à d’autres espèces cultivées en fonction des besoins de la communauté scientifique.
Ce projet est financé par un consortium de 5 sociétés privées françaises et étrangères et par l’INRAE.
FAST-NEUTRON
Pour accroitre la diversité allélique des collections de mutants sur la plateforme EPITRANS, un projet est en cours de développement pour exploiter des collections irradiées par Fast-Neutron en TILLING.
Ce type de mutagenèse est largement utilisé pour induire des mutants « perte de fonctions ». Cependant, l’utilisation de ces collections en génétique inverse est restée limité, en absence de méthodes de détection suffisamment efficaces pour identifier à la fois de grandes délétions et de petits INDELS et SNPs.
En parallèle du système haut-débit de détection de SNPs par NGS, la plate-forme développe actuellement un deuxième outil de détection spécifique des grandes délétions et des petits INDELS en s’appuyant sur 2 technologies : le NGS et la PCR digitale en gouttelettes.

Lorsque le système de détection sera validé, il pourra être élargi à d’autres espèces cultivées en fonction des besoins de la communauté scientifique.
Le projet est financé par l’INRAE et un partenaire privé.
N’hésitez pas à nous contacter si vous êtes intéressés par cette approche.
