- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Analyse à l'échelle du génome des gènes NL dans le génome du haricot commun
Equipe GDYNPATH / Valerie Geffroy
Analyses des petits ARN, de la méthylation de l'ADN et de la séquence du génome du haricot commun à l'aide de nouvelles technologies de séquençage (gènes de résistance et ADN satellites).
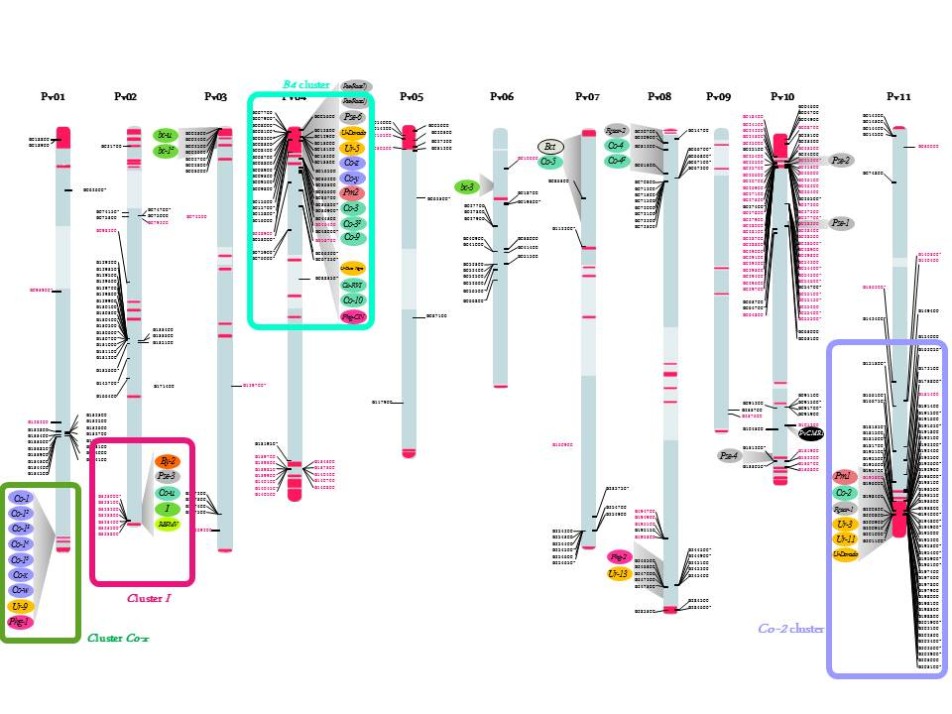
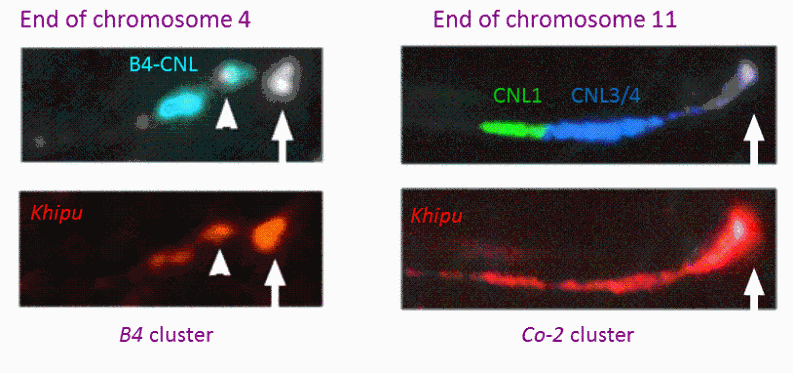
Autrefois considérée comme une espèce orpheline, d'importantes ressources génomiques sont maintenant disponibles pour le haricot commun grâce à l'avènement des nouvelles technologies de séquençage (NGS). En particulier, la séquence du génome du haricot commun, G19833 a récemment été assemblée par un consortium dirigé par S.A. Jackson (UGA ; USA) (Schmutz et al., 2014). Notre groupe a été impliqué dans ce projet par l'annotation des séquences NL. L'accès à la séquence du génome du haricot commun donne un aperçu inégalé de l'organisation et de l'évolution de cette grande famille multigénique chez P. vulgaris. Nous avons identifié ~400 NLs qui sont, comme observé chez d'autres espèces végétales, organisés en groupes complexes de gènes avec la particularité d'être souvent situés dans des régions subtélomériques près des knobs terminaux contenant l'ADN satellite khipu (Richard et al. 2013, Ribeiro et al. 2017 ; Meziadi et al. 2016 ; Chen et al. 2018) (figure 1). Des NL apparentées sur le plan phylogénétique se répartissent sur différentes extrémités chromosomiques, ce qui suggère des échanges fréquents de séquences entre chromosomes non homologues. Nos résultats soulignent donc l'importance de la recombinaison ectopique dans l'évolution des clusters de gènes R et leur distribution au sein du génome. Ces résultats confirment nos analyses précédentes basées sur l’analyse des séquences de khipu à l'échelle du génome ou des séquences NL des clusters Co-2 et B4 R montrant la plasticité des subtélomères du haricot (David et al. 2009 ; Richard et al. 2013 ; Chen et al. 2018). Cette dynamique exceptionnelle des subtélomères a été mis en évidence chez des organismes aussi divers que la levure et le parasite Plasmodium (Brown et al., 2010, Current Biol.), mais rarement dans les plantes.
La localisation particulière des séquences NL, à proximité de régions hétérochromatiques (Figure 2), nous a amenés à étudier leur statut de méthylation de l'ADN en utilisant une carte de méthylation de cytosine du génome entier (Collaboration avec le groupe S.A. Jackson, UGA, USA). Chez le haricot commun, nous avons montré que les gènes NL présentaient un profil de méthylation de l'ADN inhabituel puisque la moitié d'entre eux sont méthylés dans les trois contextes de séquence (CG, CHG, CHH), ce qui rappelle ce qui est observé pour les séquences répétées (Richard et al. 2018a). De plus, 90 NL ont également été abondamment ciblés par 24 nt siRNA, la quasi-totalité d'entre eux (95%) correspondant à des gènes NL méthylés. Cela suggère l'existence, chez le haricot commun, d'un mécanisme de silencing de ces gènes NL au niveau transcriptionnel par la voie de la RdDM (RNA-directed DNA methylation) qui n'a pas été décrite chez d'autres espèces végétales pour les gènes NL (Richard et al. 2018). Par conséquent, en plus des mécanismes bien décrits de silencing des gènes NL au niveau post-transcriptionnels impliquant des microARNm ciblant les ARNm de NL (Fei et al. 2016 ; MPMI), nos résultats suggèrent l'existence d'une couche supplémentaire de régulation des NL au niveau transcriptionnel. Ensemble, ces mécanismes pourraient être essentiels pour limiter l'expression de la résistance chez les plantes dans des conditions normales de croissance, en l'absence d'attaque pathogène, afin d'éviter les coûts associés (Richard et al. 2018b). Nous avons également identifié des microARNs de haricot ciblant les ARNm NL et déclenchant la production de petits ARNs interférents secondaires phasés (phasiRNAs) (Collaboration avec le groupe Blake Meyers ; Centre Danforth ; USA).
Nous étudions actuellement la dynamique de la méthylation de l'ADN après une infection pathogène à l'échelle du génome.
En plus de ces analyses à l'échelle du génome, nous avons un intérêt particulier pour les clusters de résistance à l'anthracnose (Co-2, Co-x, B4) et pour le cluster I de résistance aux virus (Figure 1).
