- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données

Recherche
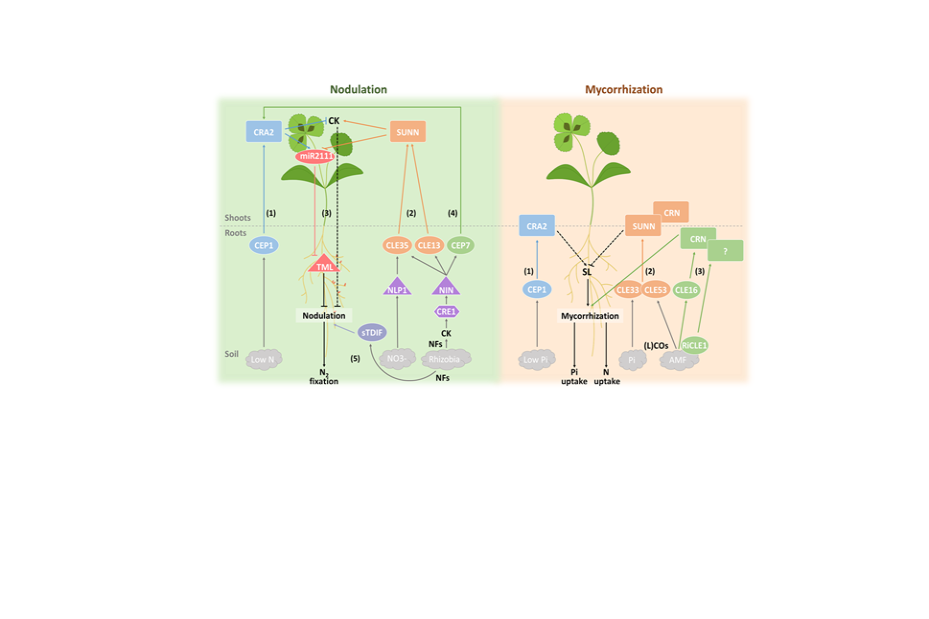
Peptides de signalisation et endosymbioses
Lire la suiteRecherche
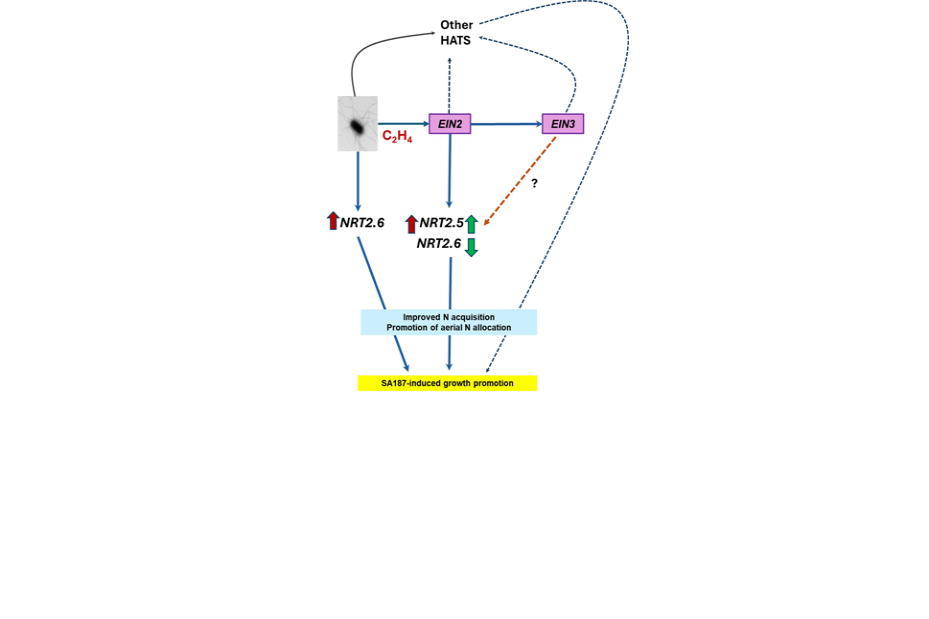
une bactérie bénéfique en faible azote
Lire la suiteRecherche
Bonne année 2026
Lire la suiteRecherche
Noël à l’IPS2
Lire la suiteRecherche
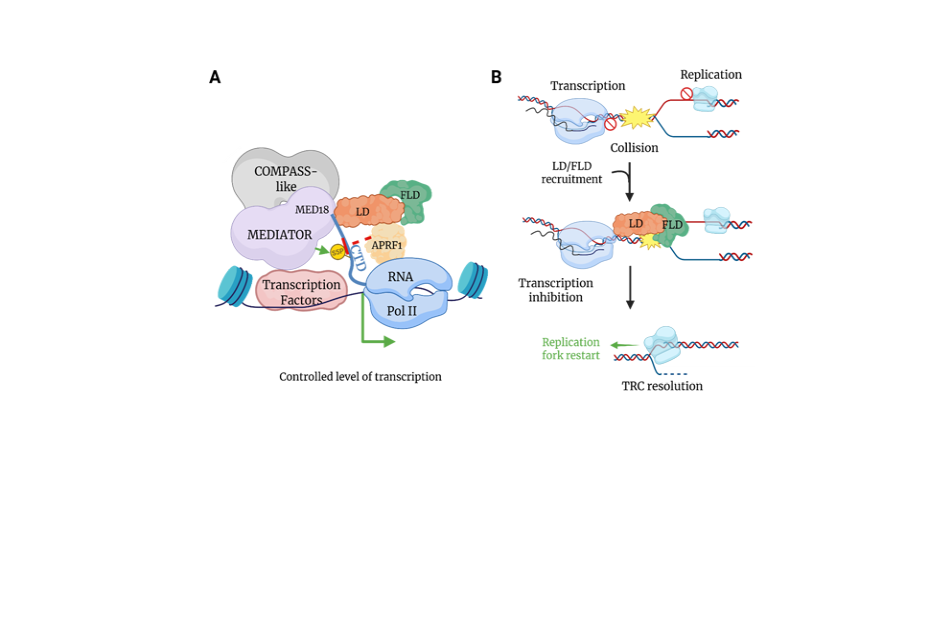
Répression globale de la transcription chez les plantes
Lire la suiteRecherche
William BOUARD a rejoint l'équipe CCARS
Lire la suiteRecherche
Un nouveau projet ERC à IPS2 !
Lire la suiteRecherche
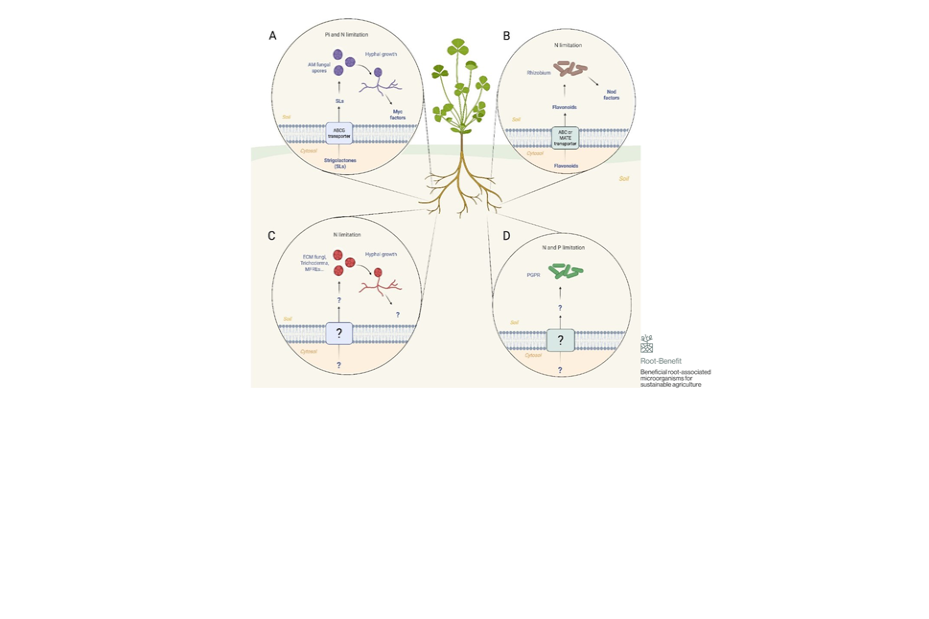
Une review sur les mécanismes moléculaires des interactions bénéfiques plantes micro-organismes
Lire la suiteRecherche
Ecole d’été “Saclay Plant Sciences” (SPS) 2026
Lire la suiteRecherche
Lancement du réseau doctoral européen HeatDDR
Lire la suiteRecherche
Transcriptomique à grande échelle et à moindre coût
Lire la suiteRecherche
ARRIVEE DE BRUNO GUILLOTIN A IPS2
Lire la suite