- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Analyses disponibles
PMM la plateforme métabolisme-métabolome de l'IPS2
Thématiques de recherche:
La Plateforme sert de support aux recherches sur les métabolismes primaires (carboné, azoté et soufré) et secondaires des plantes et leurs réponses aux contraintes environnementales. Le rôle de la Plateforme consiste en l’établissement de profils métaboliques (GC-MS, LC-MS), l’identification, la quantification des métabolites d’intérêt (RMN, LC), la détermination éventuelle de leur composition isotopique (IRMS et GC-C-IRMS) et le suivi de marquage (RMN).
Analyses GC-MS :
- Profilage des métabolites primaires
L’Analyse GC-MS permet l’identification d’un grand nombre de métabolites (bibliothèque d’environ 700 métabolites) préalablement extraits d’une matrice puis purifiés par chromatographie en phase gazeuse.
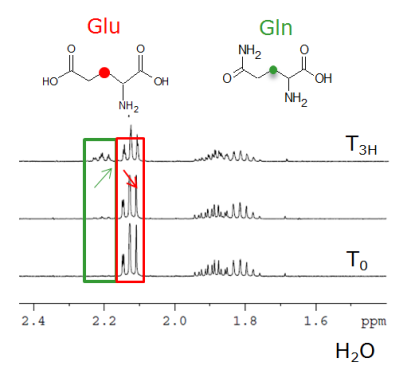
Cette technologie permet de réaliser des spectres métabolomiques de l’ensemble des composés de faible poids moléculaire (acides aminés, acides organiques, mono et disaccharides ...) et d’avoir leurs quantifications relatives. Toutes les données peuvent être traitées avec le logiciel Mev pour appliquer un traitement statistique et trouver de possibles corrélations.
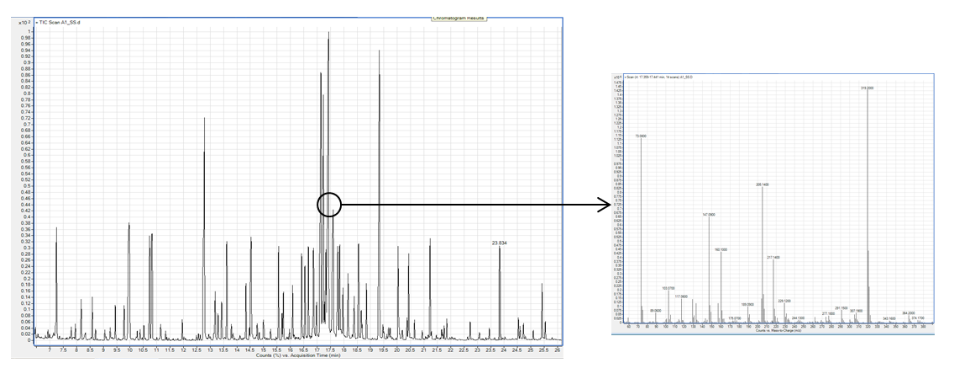
- Dosage de métabolites spécifiques.
La plateforme peut développer, à façon des analyses ciblées pour une classe de métabolites d’intérêt (terpènes, alcanes, acides gras, arômes, composés volatiles…).
Analyses LC :
Les analyses LC permettent selon l’application la quantification absolue d’une classe de métabolite. Les applications sont :
- Les acides aminés (NB la Proline ne peut pas être dosée par cette méthode)
- Les sucres (Saccharose, Glucose et Fructose)
- Les cations (Li+,Na+,NH4 +,K+,Mg+,Ca+)
- Les anions (F-, Cl-,NO2 -,Br-,NO3 -,P-,SO3 -)
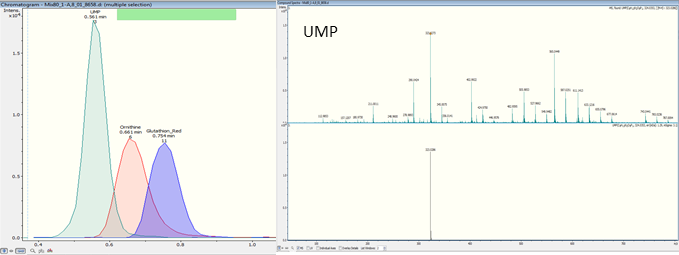
Analyses LC-MS et LC-MS-MS:
L’analyse LC-MS permet la quantification absolue de 25 cofacteurs-nucléotides préalablement extraits d’une matrice. La chromatographie liquide va permettre de séparer les différents métabolites présents dans la matrice et le spectromètre de masse de les identifier et les quantifier par la masse exacte de l’ion de chaque cofacteur-nucléotides.Les dosages de mycotoxines sont également possibles par cette technique.
Analyses IRMS :
La Plateforme possède deux spectromètres de masse isotopique (IRMS) et plusieurs systèmes couplés permettent d’obtenir un gaz pur à partir d’échantillons solides, liquides et gazeux. Les spectromètres de masse isotopiques mesurent le rapport isotopique (13C/12C, 15N/14N, 18O/16O ou encore D/H) du gaz pur (CO2, N2, O22) issu d’échantillons solides, liquides ou gazeux et le comparent à celui d’un gaz de référence (étalonné par rapport au standard international).
- Couplage EA-IRMS :
Ce couplage est destiné à l’analyse isotopique du carbone (13C/12C) et de l’azote (15N/14N) de la matière organique totale (poudre sèche végétale ou animale) ou de métabolites préalablement purifiés par HPLC (ex. acides aminées, acides organiques, sucres solubles).
- Couplage GC-C-IRMS et Couplage LC-co-IRMS :
Ces couplages permettent l’analyse isotopique de composé-spécifique préalablement extraits (feuilles de plantes par exemple) et séparés par des techniques de chromatographies (liquide ou gazeuse)
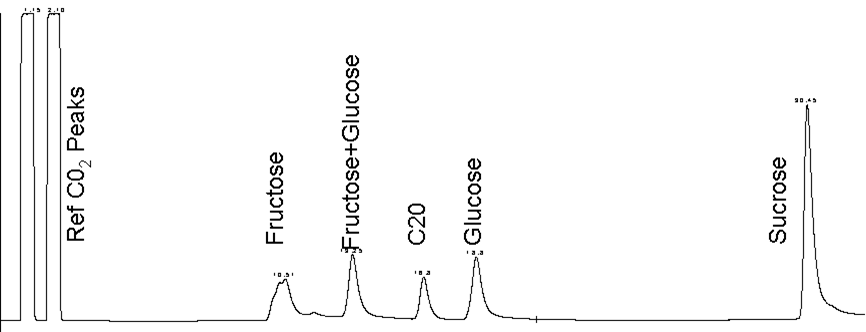
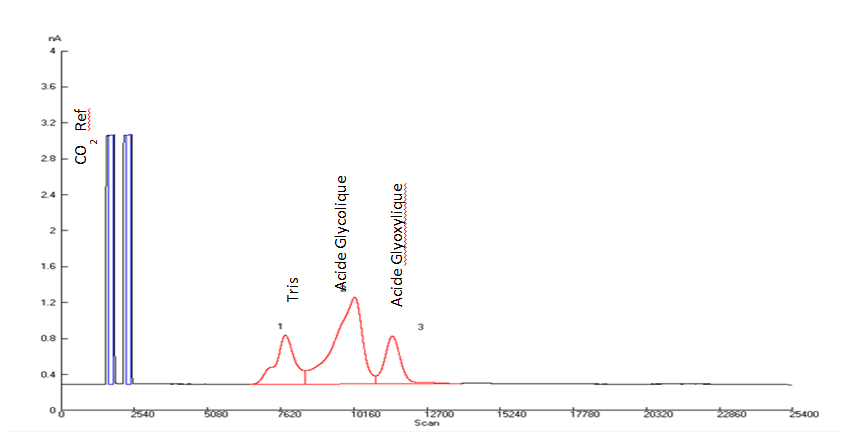
Analyses RMN :
Les analyses RMN permettent l’acquisition d’expériences 1 Dimension : H, 13C, 15N et 31P et/ou 2 Dimensions homo ou hétéro nucléaire (ex : HSQC, COSY….).Ces acquisitions peuvent permettent la quantification absolue dans la matrice (ex : Activité enzymatique), l’identification structurale ou encore de pouvoir de suivre un enrichissement isotopique dans les positions intramoléculaires.