- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Plateforme Interactomique
Les interactions entre protéines sont des éléments essentiels des systèmes biologiques et leur analyse peut fournir des indications précieuses sur les fonctions des protéines.
À ce jour, le système double-hybride chez la levure (Y2H) est seule méthode permettant la détection des interactions protéine-protéine à haut débit in vivo. L'équipe « Expression des Génomes des Organites – OGE » de l’IPS2 a été impliquée dans le projet international « Arabidopsis Interactome Mapping » (1) coordonné par le Pr Joseph Ecker (Salk Institute, San Diego) et le Pr Marc Vidal (DFCI, Boston). Cette première carte du réseau des interactions protéiques chez Arabidopsis a été générée grâce à l’utilisation de la technique de Y2H développée au DFCI pour C. elegans, l’homme et la levure (2). Les premières données de ce projet ont été publiées en 2011 (AIM, 2011) et correspondent à environ 6200 interactions entre 2700 protéines. L’ensemble de ces données est accessible à la communauté scientifique : Plant Interactome Database .
Récemment, l'équipe OGE a entièrement amélioré et automatisé le protocole Y2H qui a été précédemment utilisé pour le projet AIM. Grâce à l’utilisation de plaques 384 puits, à un protocole entièrement en milieu liquide et à l’utilisation de robots, la plate-forme SPOmics-Interactome est capable de cribler un pool de 50 protéines hybrides (DB- X) contre la banque de protéines hybrides AD-AIM (environ 12000 protéines d’Arabidopsis) en deux mois.
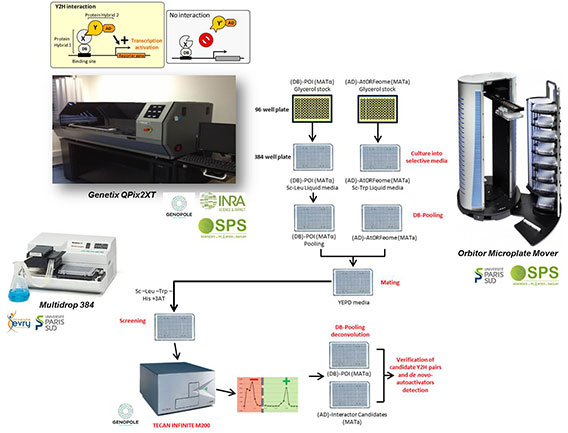
Les plasmides portant les ORFs codant pour les protéines d’intérêt fournis par nos collaborateurs dans le vecteur pDEST - DB employé sur la plateforme sont utilisés pour transformer des levures puis les protéines DB-X hybrides sont testées pour éliminer celles capables d’auto-activation. Les levures exprimant les protéines DB-X hybrides sont cultivées individuellement dans des milieux sélectifs, puis combinées en pools de 50 clones et criblées contre la banque AD-AIM. Après identification des protéines candidates, une analyse matricielle en Y2H est effectuée par « déconvolution » des 50 DB-proies et par tests individuels contre les candidats AD-appâts. Une étape finale de séquençage des protéines AD- et DB- permet la validation de l’identité des protéines en interaction.
Chaque nouvel ORF criblé est intégré dans la banque AD- permettant une constante implémentation du réseau.
Prix (académique) : 12,500 euros pour un crible d’un maximum de 50 protéines DB-X.
(1) Evidence for Network Evolution in an Arabidopsis Interactome Map. Arabidopsis Interactome Mapping Consortium. Science 29 Jul 2011: Vol. 333, Issue 6042, pp. 601-607
(2) High-Quality Binary Interactome Mapping. Dreze M, Monachello D, Lurin C, Cusick ME, Hill DE, Vidal M, Braun P. Methods in enzymology 470:281-315 January 2010
