- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Les ARN non-codants : des acteurs de la plasticité développementale de la racine
Équipe REGARN / Martin Crespi
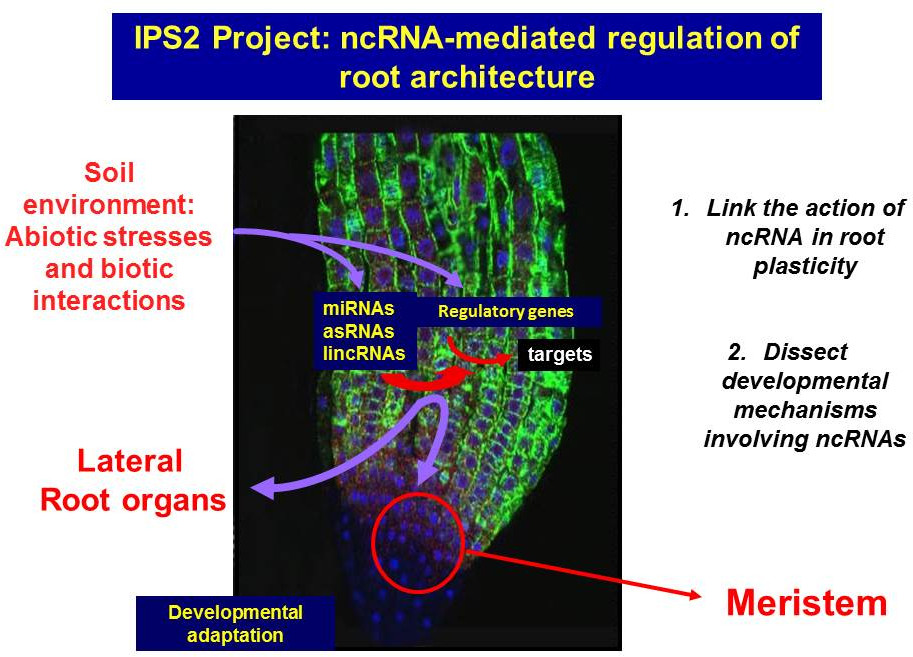
Objectifs scientifiques et stratégie
Les approches génomiques ont changé notre vision du transcriptome eucaryote. L’une des principales découvertes liées à ce séquençage à grande échelle est que le nombre de transcriptions non codantes de protéines, généralement appelées ARN non codants (ou ARNnc) est beaucoup plus élevé qu’on ne le croyait auparavant, y compris les microARN, les siARN interférents de petite taille et le nombre élevé de longs ARNnc (intergéniques ou antisens, Ariel et al., 2015). Au cours des dernières années, il a été démontré que de nombreux longs ARNnc agissent comme régulateurs quantitatifs positifs ou négatifs de l’expression génique. La variation des séquences nucléotidiques ou des modèles d’expression du génome non codant peut avoir moins d’effets pléiotropes que les changements dans la séquence protéique des régulateurs critiques. Ainsi, outre les promoteurs, les introns et les transposons, des régions englobant des ARN non codants apparaissent comme des acteurs de l’adaptation des plantes aux contraintes environnementales.
M. Crespi / Équipe REGARN
Les mécanismes de développement impliquant des ARNnc sont étudiés activement. Les miARNs et les siARNs jouent des rôles essentiels chez les eucaryotes en guidant le clivage de l’ARNm cible ou la répression translationnelle après intégration dans un complexe ribonucléoprotéique, le RISC (« RNA Silencing Induced Complex »). En effet, les miARNs et certains siRNAs participent à des boucles de régulation complexes avec leurs cibles (généralement des facteurs de transcription) pour contrôler plusieurs aspects du développement racinaire (ex. Lelandais-Brière et al., 2016). En revanche, l’action des ARNnc et de la majorité des siRNAs est beaucoup moins connue (Ariel et al., 2015). Le développement et la croissance des racines sont des processus complexes intégrant une grande variété de voies de signalisation (endogènes comme les hormones ou exogènes comme les nutriments ou la teneur en eau du sol), ce qui donne des architectures racinaires différentes. Ainsi, la racine est capable d’ajuster sa croissance et son développement en fonction des conditions environnementales. Modifier spatialement ou temporellement l’action des miARNs ou d’autres ARNnc régulateurs est une façon pour les plantes d’intégrer les signaux de l’environnement dans leur croissance et leur développement futurs. Un autre modèle de plasticité racinaire est la capacité des légumineuses à interagir avec des symbiotes bactériens pour développer de nouveaux organes, les nodules fixant l’azote. Les nodules racinaires sont de nouveaux organes où l’expression de milliers de gènes est reprogrammée dans les deux symbiotes, la bactérie et la cellule hôte végétale, dans un contexte de développement défini. C’est donc un bon modèle pour comprendre le rôle des ARN régulateurs dans l’organogenèse.
Notre projet vise à comprendre le rôle des ARNnc dans la plasticité racinaire. À l’IPS2, notre laboratoire développe deux axes de recherche utilisant Arabidopsis thaliana et Medicago truncatula pour répondre à des questions clés émergentes dans le domaine des ARN non codants :
- Établir un lien entre les ARNnc et la plasticité racinaire en explorant la diversité des génomes et des écotypes.
- Disséquer les mécanismes du développement déclenchés par les ARNnc dans les processus de développement racinaire.