- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Exploration de la diversité des ARNnc : lier les ARNnc à la plasticité racine
Équipe REGARN / Martin Crespi
Dans ce premier thème, nous avons utilisé une série d’approches génomiques pour identifier et annoter les longs et petits ARNnc, principalement chez M. truncatula et A. thaliana et plus récemment chez d’autres espèces en collaboration avec différents consortiums, afin de détecter des ARNnc potentiels associés aux changements dans la réponse de croissance racinaire. Nous avons réalisé une analyse globale des mi/siARN dans M. truncatula (conservés et nouveaux, spécifiques des légumineuses ou seulement de Medicago ) dans le cadre du projet MIRMED (coordinateur M. Crespi) où 23 conditions différentes de croissance de racines ont été utilisées. Cela nous a permis d’analyser les réseaux de petits ARN dans cette légumineuse modèle, y compris de nouvelles cibles de miARN conservés ainsi que de nouveaux miARN, notamment dans le contexte de la symbiose (Formey et al, 2014, Lelandais-Brière et al., 2016). Nevin Young (et des collaborateurs internationaux, dont J. Gouzy, F. Débelle et L. Gentzbittel en France) développent actuellement une connaissance approfondie des génomes des accessions de M. truncatula. Il est intéressant de noter que des études d’association à l’échelle du génome (GWAS) réalisées dans le laboratoire de N. Young pour identifier de nouveaux gènes impliqués dans l’adaptation de la symbiose à différents environnements ont montré que la majorité des loci identifiés correspondent à des régions intergéniques. Plus récemment, nous avons participé à un projet de collaboration visant à utiliser la technologie PACBIO pour le séquençage détaillé du génome de M. truncatula qui a permis la détection « d’îlots symbiotiques » où un grand nombre de long ARNnc ont été observés comme « intégrés » dans des réseaux de corégulation avec des gènes de symbiose (Pécrix et al, Nature Plants sous presse). Nous avons également analysé la diversité miRNA/siRNA dans le cadre d’un vaste projet collaboratif sur le tournesol lié au séquençage complet de ce génome géant (Badouin et al., 2017).
En plus de leur identification, nous avons commencé l’analyse fonctionnelle de certains miARN chez M. truncatula. Nous avons pu montrer que miR160 et miR396 régulent la croissance racinaire, la réponse à l'auxine et la mycorhization. Nous avons mis en évidence l’impact de RDR6 et des phasiARN associés dans de nombreux aspects du développement des plantes (Bustos-Sanmamed et al, 2013, Bazin et al, 2013, Bustos-Sanmamed et al, 2014). Plus récemment, nous avons analysé l’action de nouveaux miARNs identifiés comme étant exclusivement exprimés dans l’apex racinaire (Formey et al., 2014). L’inhibition de l’action de l’un de ces miARN a fortement empêché la répression de la croissance racinaire par le cadmium. Ce miARN coupe un long ARNnc pour produire des phasiARN secondaires qui peuvent cibler des gènes spécifiques limitant la croissance racinaire en réponse au cadmium dans les phénotypes sensibles, comme nous avons pu le démontrer en testant le mutant rdr6 altéré dans les phasiARN mais pas dans la voie tasiARN (Bustos-Sanmamed et al., 2014). Le fait que ce nouvel miARN a également été différentiellement induit entre des écotypes présentant des réponses de croissance racinaire contrastées au cadmium renforce encore un lien potentiel avec l’adaptation des écotypes. Nous proposons que ce réseau de régulation impliquant un nouveau miARN et des longs ARNnc ciblés produisant des phasiARN, module la réponse des racines de Medicago au cadmium, un trait très intéressant (Proust et al., en préparation).
Chez Arabidopsis thaliana, nous avons concentré nos efforts sur l’adaptation des racines à la carrence phosphate. Chez cette espèce, une faible concentration de phosphate inorganique (faible-Pi) réduit la croissance des racines primaires au profit des racines latérales.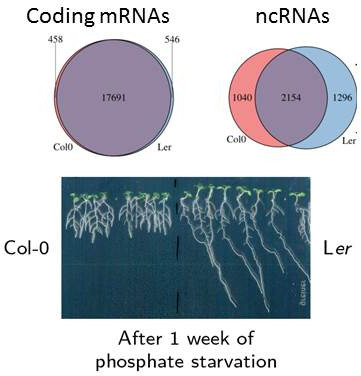
Fait intéressant, une approche QTL a révélé les gènes LPR (LOW PHOSPHATE RESPONSIVE) dont l’expression dans la coiffe racinaire est nécessaire pour une faible réponse Pi. Ces données et d’autres suggèrent que la détection de Pi à l’apex de la racine est essentielle pour modifier l’architecture racinaire pendant la carrence phosphate. Nous avons commencé, en 2013, une collaboration avec le laboratoire de L. Nussaume (CEA, Cadarache, France) pour étudier l’évolution des réseaux d’ARNnc dans l’apex racinaire des écotypes (Col-0 et Ler) montrant des réponses différentielles de croissance racinaire à faible Pi (projet ANR RNADAPT). En utilisant le séquençage d’ARN, nous avons comparé la réponse transcriptionnelle complète (ARNm, long ARNnc et petits ARN) des extrémités des racines de ces deux écotypes. Nous avons identifié des milliers de nouveaux longs ARNnc conservés au niveau de l’ADN. Cependant, contrairement aux gènes codants, de nombreux long ARNnc étaient spécifiquement transcrits dans un écotype (voir la figure montrant le nombre d’expression spécifique à l’écotype des ARNm et des ARNnc codants). Ces longs ARNnc spécifiques d’écotypes ont été caractérisés par l’analyse de leur variabilité parmi les écotypes séquencés d’Arabidopsis et de leur capacité à générer des siRNA. La majorité des longs ARNnc étaient régulés différemment en fonction du génotype plutôt que de la carence phosphate, ce qui suggère un rôle dans l’adaptation des écotypes. Nous avons effectué plusieurs cribles (comme des modèles de corégulation avec les gènes voisins ou des interactions antisens avec des régulateurs critiques connus) pour lier ces longs ARNnc spécifiques d’écotypes à la réponse différentielle de croissance racinaire entre écotypes. L’analyse fonctionnelle a révélé que la mauvaise expression de deux longs ARNnc spécifiques à l’écotype affecte la croissance racinaire primaire du Col-0 (Blein et al., en préparation). Concernant les miARN, nous avons poursuivi l’analyse de l’action des miARN sur des facteurs de transcription spécifiques impliqués dans l’architecture racinaire tels que miR169 et NFYA2. Nous avons démontré que des isoformes miR169 spécifiques, portant des mutations ponctuelles par rapport à la séquence miR169 canonique, étaient impliquées dans la formation des racines latérales et la croissance des racines primaires (Sorin et al., 2014).
Une autre question émergente dans le domaine des ARNnc est de savoir si ces nouveaux ARNnc sont vraiment non codants ou s’ils peuvent coder pour de petits peptides ? Pour répondre à cette question, nous avons établi une collaboration avec Julia Bailey Serres (UCSF Riverside, USA) dans le cadre d’une bourse Marie Curie sortante. Nous avons déterminé l’empreinte ribosomique des ARN polyadénylés dans les racines d’Arabidopsis, qui ont été transférés de Pi élevé à Pi faible. Nous avons trouvé, sur 2382 longs ARNnc potentiels, que près de la moitié étaient associés aux ribosomes. La prédiction des petits ORF (sORF), le calcul de la terminaison des ribosomes et l’analyse des peptides ont révélé que de nombreux sORF sont cachés dans les longs ARNnc et peuvent produire de petits peptides. En effet, nous avons identifié 34 peptides conservés au cours de l’évolution et régulés conditionnellement, codés dans des ARNnc. De plus, dans le cas des transcriptions antisens naturelles (NAT), les sORF avec empreintes de ribosomes prédominaient près des extrémités 5’ des transcripts. Il s’agit notamment des NATs régulés par le Pi, dont l’activation est corrélée avec l'augmentation de la traduction de l’ARNm sens. De plus, nos données fournissent des preuves de la traduction de long ARNnc bien connus comme les siARN trans-acting et phasés (tasiARN et phasiARN) et une éponge à miRNA cible mais pas de précurseurs de miARN. Grâce à des analyses de mutations, nous avons montré que la traduction d’une sORF non conservée dans le gène TAS3a améliore la biogénèse des tasiARN apparentés. Dans l’ensemble, nous proposons que la traduction des sORF puisse déterminer la stabilité et la fonction de certains ARNnc (Bazin et al., 2017). Les ensembles de données développés dans ce contexte nous ont permis de classer plus en détail les longs ARNnc mentionnés précédemment (lorsqu’ils sont détectés dans Col-0) en fonction de leur association aux ribosomes.
Parallèlement à ces travaux, nous avons poursuivi l’étude de l’interaction entre les mécanismes de dégradation par les mi/siARN et ceux impliqués dans le contrôle de qualité des ARNm (RQC ou contrôle de qualité des ARNm ; Moreno et al., 2013). Nous avons montré que les « P-bodies » et les « siRNA-bodies » agissent de manière antagoniste sur des ARN « aberrants » similaires. Ces « bodies » adjacents ont révélé de nouveaux aspects de la dynamique subcellulaire de la dégradation des ARN (Martinez-Alba et al., 2015). Nous étendons ce travail en utilisant plusieurs mutants du RQC et d’épissage (dans le fond Col-0) pour évaluer la dépendance de l’expression des longs ARNnc dans les écotypes (par exemple, l’écotype Ler porte une mutation du gène SOV impliqué dans le contrôle qualité des ARN, RQC). Cela pourrait nous permettre d’explorer davantage les liens entre l’évolution de nouvelles voies de régulation dans les écotypes par le biais de mutations dans les voies de RQC ou d’épissage.
