- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Le clonage positionnel
Le clonage positionnel a été développé sur la plateforme EPITRANS dans le cadre d’une approche de génétique classique sur des collections de mutants induites à l’EMS ou des collections d’accessions naturelles chez les plantes cultivées. Cette approche permets de valoriser le matériel biologique crée par l’INRA en clonant et en caractérisant des gènes d’importances agronomiques susceptibles d’être utilisés en sélection.
En effet, plusieurs collections de mutants ont été phénotypées afin qu’elles puissent être valorisables en génétique classique et en génétique inverse. Pour organiser ces données et les rendre accessibles à la communauté scientifique, la plateforme a créé la base de données UTILLdb (Dalmais et al., 2008) qui recense tous les phénotypes, hiérarchisés par traits, des collections affichées dans la base (pois, tomate, Brachypodium et Lin) Cette ressource est ouverte à tous et les graines de lignées d’intérêts peuvent être demandées via les personnes à contacter.
La technologie utilisée sur la plateforme pour cloner des gènes d’intérêts agronomiques est l’analyse en pools de ségrégeants (BSA) par séquençage NGS (MiSeq, Illumina, CLC sequence viewer).
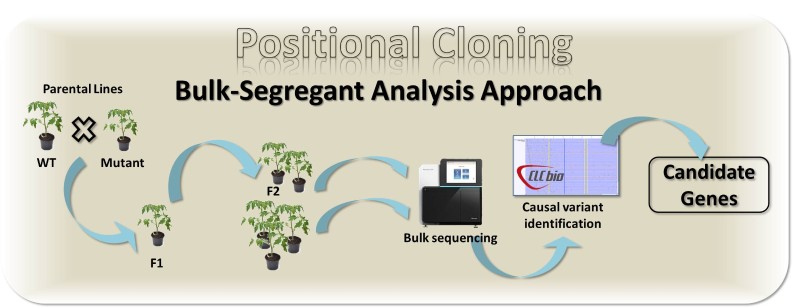
En fonction des objectifs, du matériel et des moyens de nos partenaires, la plateforme propose son expertise à différents niveaux, pouvant être combinés :
- Criblage d’une collection de mutants induite chimiquement pour un trait phénotypique donné.
- Séquençage des pools de ségrégants suivi de l’analyse bio-informatique pour identifier le ou les régions causales.
- Cartographie génétique fine et validation fonctionnelle du ou des gènes candidats identifiés.
Actuellement, une vingtaine de gènes d’intérêts agronomiques ont été clonés sur la plateforme dont des gènes de résistance aux pucerons (Vat), aux virus (nsv, pvr2, ZYM, etc.), aux champignons (Fo2, PmW, etc.), aux bactéries (Bs), 4 gènes du déterminisme du sexe chez les Cucurbitacées (M, G, A and A2), 1 gène de la restauration de la fertilité mâle (Rfo) et des gènes de floraison et de développement (Ti, Rms2, HR, J-2, etc.)
Sept brevets ont été déposés et plus de 40 articles ont été publiés.
Articles majeurs :
Ligerot Y, de Saint Germain A, Waldie T, Troadec C, Citerne S, Kadakia N, Pillot JP, Prigge M, Aubert G, Bendahmane A, Leyser O, Estelle M, Debellé F, Rameau C. (2017) The pea branching RMS2 gene encodes the PsAFB4/5 auxin receptor and is involved in an auxin-strigolactone regulation loop. PLoS Genet. Dec 8;13(12):e1007089. doi: 10.1371/journal.pgen.1007089. eCollection 2017 Dec.
Boualem A, Troadec C, Camps C, Lemhemdi A, Morin H, Sari MA, Fraenkel-Zagouri R, Kovalski I, Dogimont C, Perl-Treves R, Bendahmane A. (2015). A cucurbit androecy gene reveals how unisexual flowers develop and dioecy emerges. Science. 350(6261):688-91. doi : 10.1126/science.aac8370.
Martin A, Troadec C, Boualem A, Rajab M, Fernandez R, Morin H, Pitrat M, Dogimont C and Bendahmane A (2009) A transposon-induced epigenetic changes leads to sex determination in melon. NATURE 461:1135-1139
Boualem A, Fergany M, Fernandez R, Troadec C, Martin A, Morin, Sari M, Collin F, Flowers M, Pitrat M, Purugganan M, Dogimont C, Bendahmane A (2008) A Conserved Mutation in an Ethylene Biosynthesis Enzyme Leads to Andromonoecy in Melons. Science 321:336-338.
