- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Développement et mise en œuvre de VIGS pour le haricot commun et le pois à l'aide du BPMV
Équipe GDYNPATH / Valérie Geffroy
Développement et mise en œuvre due VIGS (Virus-Induced Gene Silencing) chez le haricot commun et le pois (Pisum sativum) en utilisant le BPMV (Bean pod mottle virus)
Malgré la séquence du génome et d'autres ressources -omiques qui sont maintenant disponibles pour le pois et le haricot commun, la caractérisation fonctionnelle des gènes est encore limitée chez ces deux espèces. En effet, comme la plupart des légumineuses, elles sont toutes deux récalcitrantes à une transformation génétique stable. Dans ce contexte, le VIGS est un outil intéressant de génomique fonctionnelle.
Le VIGS permet une analyse rapide et transitoire de la fonction des gènes chez les légumineuses qui sont difficiles à transformer. Le VIGS exploite la réponse de défense naturelle des plantes contre les virus, à savoir le PTGS (Post-Transcriptional Gene Silencing) qui cible les ARN viraux et conduit à une dégradation séquence-spécifique. Cette technologie implique le développement de vecteurs viraux recombinants portant dans leur génome un fragment de séquence homologue à un gène endogène d'intérêt. Après infection par ce virus recombinant, les transcrits du gène d'intérêt de la plante deviendront des cibles de dégradation par le mécanisme PTGS, conduisant ainsi à son extinction transitoire.
Nous avons choisi de travailler avec le vecteur VIGS dérivé du BPMV parce que c'est actuellement le vecteur VIGS le plus largement utilisé chez les légumineuses (Pflieger et al. 2013). De plus, il est adapté aux études VIGS à haut débit car l'infection des plantes se fait en une seule étape par inoculation mécanique des feuilles à l'aide de plasmides infectieux portant le génome recombinhant du vecteur VIGS sous contrôle d'un promoteur CaMV 35S (Zhang et al. 2010 ; Plant Phys). Dans ce contexte, nous avons adapté avec succès le VIGS en utilisant le vecteur VIGS dérivé du BPMV chez le haricot commun (Pflieger et al. 2014) et le pois (Meziadi et al. 2016).
● VIGS chez le haricot commun (Phaseolus vulgaris)
Des études antérieures ont montré que le vecteur viral dérivé du BPMV , un vecteur VIGS largement utilisé chez le soja (Glycine max), convient également à l'extinction de gènes chez Phaseolus vulgaris cv. Black Valentine (Zhang et al. 2010 ; Diaz-Camino et al. 2011).
Le succès du VIGS repose sur la capacité des vecteurs viraux à infecter le génotype d'intérêt. Contrairement au soja pour lequel presque tous les cultivars commerciaux sont sensibles à l'infection par le BPMV, seuls quelques génotypes de haricot commun (dont ‘Black Valentine’) sont sensibles au BPMV. Avec la récente disponibilité de la séquence du génome de Phaseolus vulgaris (Schmutz et al. 2014), la technologie VIGS ouvre de nouveaux horizons en génomique fonctionnelle pour évaluer les fonctions des gènes impliqués dans des caractères d'importance agronomique (résistance aux maladies, tolérance au stress abiotique…).
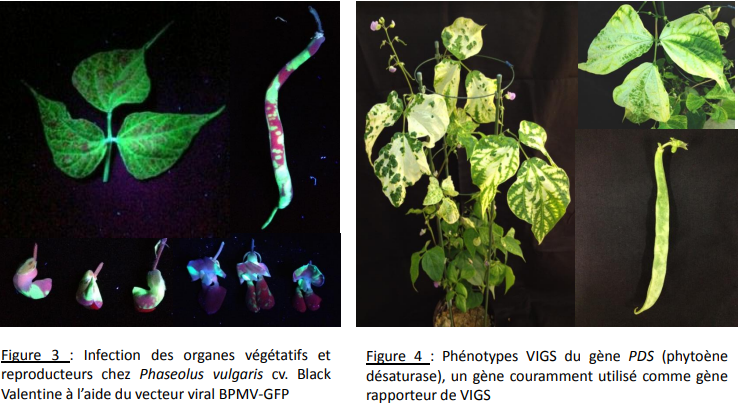
Nous avons développé un protocole de VIGS haut débit chez Phaseolus vulgaris en utilisant un vecteur viral dérivé du BPMV (Pflieger et al. 2014). La sensibilité des génotypes de haricot commun d'intérêt a été testée à l'aide du vecteur BPMV taggé GFP (Green Fluorescent protein) (Figure 3). Dans les génotypes sensibles, l'efficacité du VIGS a ensuite été testée en utilisant le vecteur BPMV-Phytoène désaturase (BPMV-PDS) (Figure 4).
● VIGS chez le pois (Pisum sativum)
Dans le cadre du projet PeaMUST (Pea MUlti-STress adaptation and biological regulations for yield improvement and stability ; projet ANR-11-BTBR-0002), nous développons l’outil VIGS à haut débit pour la validation rapide de gènes candidats impliqués dans les différents caractères agronomiques (résistance aux maladies, tolérance au gel...) chez le pois.
Nous utilisons un vecteur viral dérivé du BPMV, optimisé chez le soja et Phaseolus vulgaris. L'efficacité du VIGS dans les parties aériennes (feuilles) a été testée en utilisant le vecteur BPMV-PDS (Figure 5) et dans les racines en utilisant un vecteur BPMV-KOR1 ciblant l'endogène KORRIGAN-1 précédemment démontré comme un bon gène rapporteur du VIGS dans les racines (Constantin et al. 2004) (Figure 6).
● Validation fonctionnelle de gènes candidats dans les légumineuses à l'aide de VIGS
Nous utilisons le VIGS pour la validation fonctionnelle de gènes issus de nos propres programmes de recherche (par exemple, des gènes R candidats), mais aussi pour établir des collaborations sur des sujets de recherche sélectionnés. Par exemple, en collaboration avec le groupe de Phillip McClean (North Dakota State University ; USA), nous avons réalisé la validation fonctionnelle par VIGS d’un gène candidat pour le gène P (pigment) (McClean et al. 2018).
