- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Analyse différentielle de données ARN-seq à la paire de bases
DiffSegR : une méthode d'analyse de l'expression différentielle pour des données d’ARN-seq utilisant la détection de ruptures
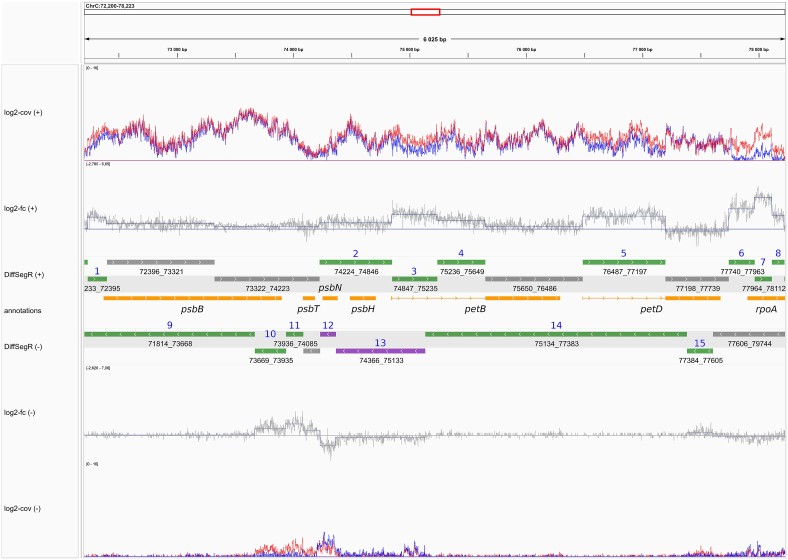
Pour comprendre pleinement la régulation des gènes, il est nécessaire d'avoir une compréhension approfondie à la fois du transcriptome et des activités enzymatiques et de liaison à l'ARN qui le façonnent. Bien que l’avènement de l’ARN-Seq soit allé de pair avec le développement de nombreux outils dédiés à l’analyse du transcriptome, la plupart de ces outils ne considèrent que l'abondance des lectures de séquençage le long de motifs annotés (tels que les gènes). Malheureusement, ces annotations sont très souvent incomplètes, ce qui conduit à des erreurs dans les analyses d'expression différentielle.
Pour répondre à ce problème, les équipes OGE et GNET de l'IPS2, avec le soutien du Phenoscope, de la plateforme POPS et du LaMME, ont publié dans NAR Genomics and Bioinformatics DiffSegR, un package R qui permet de découvrir les différences d'expression à l'échelle du transcriptome entier entre deux conditions biologiques en utilisant des données d'ARN-Seq. DiffSegR ne nécessite pas d'annotation préalable et utilise un algorithme de détection de ruptures pour identifier les limites des régions exprimées de manière différentielle en analysant le log2 du rapport entre les niveaux d'expression des deux conditions étudiées par paire de base. En quelques minutes de calcul, DiffSegR a pu prédire avec justesse les rôles des ribonucléases chloroplastiques Mini-III et PNPase dans la maturation de l'ARNr et la dégradation en 3' et en 5' des précurseurs d'ARNm, d'ARNt et des introns excisés. Nous pensons que DiffSegR sera bénéfique pour les biologistes utilisant la transcriptomique car il permet d'accéder à des informations provenant d'une couche du transcriptome négligée par les « pipelines » d'analyse de l'expression différentielle classiquement utilisés aujourd'hui. DiffSegR est disponible à l’adresse ici : https://aliehrmann.github.io/DiffSegR/index.html.
11/03/2024
