- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
ARNs non codants et adaptation à l’environnement
Le transcriptome non-codant de deux écotypes d’Arabidopsis suggère de nouveaux mécanismes d'adaptation des plantes à l'environnement du sol.
Les gènes codant pour des protéines sont largement conservés parmi les organismes. Cette conservation est encore plus forte entre les écotypes de la même espèce malgré le fait qu’ils peuvent réagir différemment à leur environnement. La partie non codante du génome est bien plus faiblement conservée et peut donc contribuer de manière significative à cette variation du phénotype, comme une catégorie particulière d'ARN qui ne codent pas pour des protéines: les ARN non codants (ARNnc). Sachant que les ARNnc régulent quantitativement l'expression des gènes codants, ils pourraient être de nouveaux acteurs clés dans la réponse à l'environnement. Chez les plantes, le développement du système racinaire est très plastique et modifie sa forme en fonction de la disponibilité en nutriments. Par exemple, le phosphate inorganique s'accumule préférentiellement dans la couche supérieure du sol, et lors d’une carence en phosphate certaines plantes arrêtent la croissance de leur racine principale pour développer des racines latérales et explorer le sol horizontalement.
L'équipe de Martin Crespi à l'Institut des Sciences du Végétal Paris Saclay (IPS2, Université Paris-Saclay), en collaboration avec les groupes de Thierry Desnos (UMR7265 SAVE, CEA Cadarache) et de Daniel Gautheret (I2BC, Université Paris-Saclay), a étudié comment les ARNnc peuvent contrôler l'adaptation des écotypes à l'environnement (10.1104/pp.20.00446). Ils ont utilisé deux écotypes d'Arabidopsis thaliana qui réagissent différemment à la carence en phosphate: a) Columbia, qui arrête la croissance de sa racine principale et b) Landsberg erecta qui continue de croître. Pour ces deux écotypes, ils ont étudié dans la racine les variations du transcriptome complet incluant les gènes codants, les ARNnc et les petits ARN, en réponse à une courte carence en phosphate. Des milliers d'ARNnc non caractérisés auparavant ont été identifiés, dont la séquence d’ADN est hautement conservée entre les deux écotypes. Malgré cette forte conservation de séquence, la moitié des ARNc détectés ne sont transcrits que dans un des deux écotypes. En revanche, 90% des gènes codant pour des protéines sont exprimés de manière équivalente dans les deux écotypes en réponse à ce stress. À l'aide de mutants ayant des niveaux d'expression modifiés de cinq nouveaux ARNnc spécifique d’un des deux écotypes, ils ont pu identifier deux ARNnc régulant la croissance des racines primaires. Un de ces ARNnc est lié à la régulation de gènes impliqués dans le contrôle de la croissance des racines et du transport des nutriments. Ainsi, en explorant les transcriptomes de différents écotypes en incluant les ARNnc, de nouveaux mécanismes de régulation impliqués dans les adaptations du système racinaire à l’environnement ont pu être caractérisés.
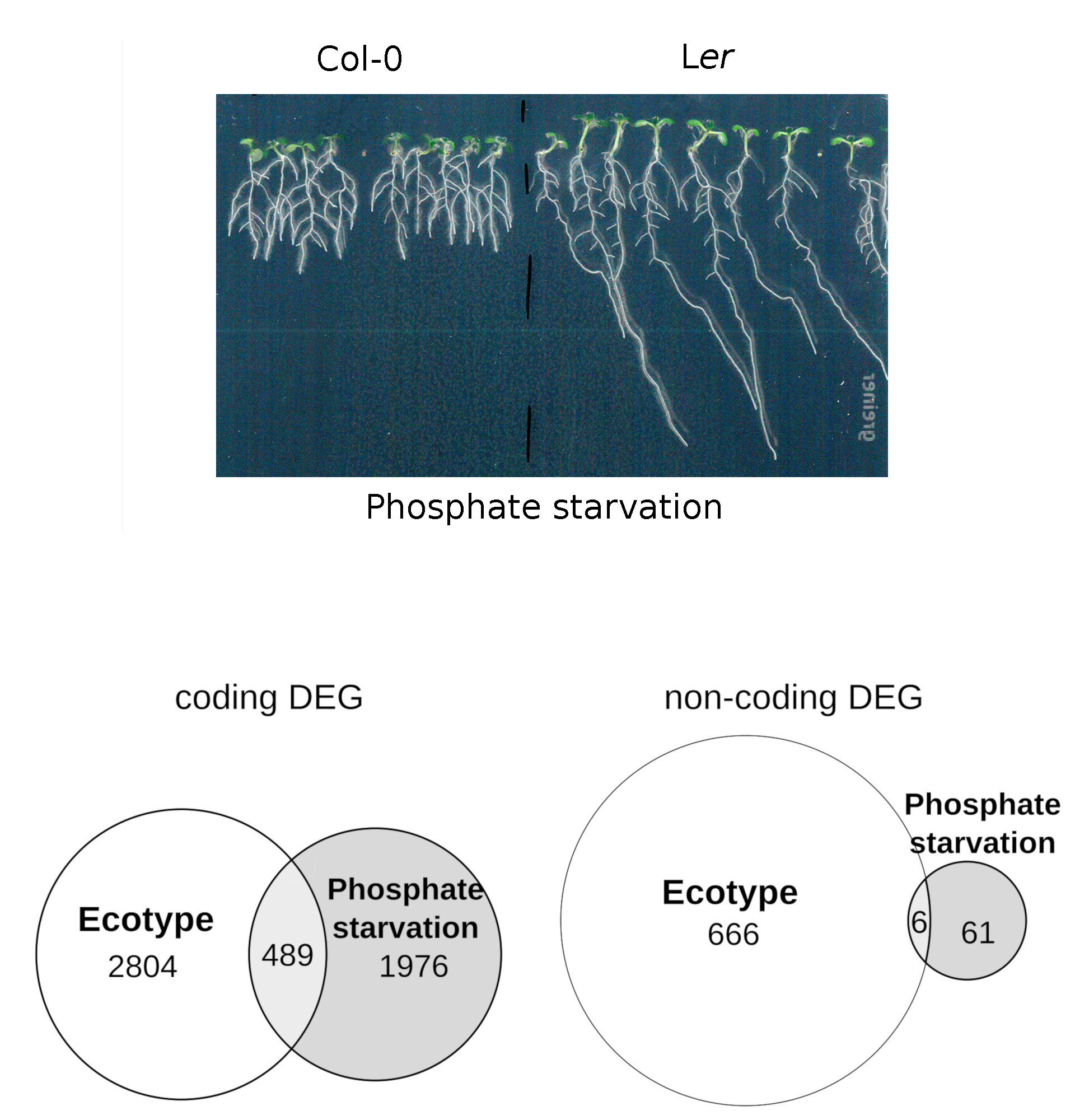
Légende de la figure : Au-dessus : Les écotypes Col-0 et Ler réagissent différemment à la privation de phosphate. En-dessous : Différences transcriptomiques entre les deux écotypes ou en réponse à un déficit en phosphate. Alors que pour les gènes codants le nombre de gènes différentiellement exprimés entre les deux écotypes (3293) ou en réponse à la carence en phosphate (2465) est du même ordre de grandeur, les gènes non codants sont clairement biaisés vers la régulation selon les écotypes (672), dont une centaine d'ARNnc "spécifiques" des écotypes (aucune expression dans l'autre écotype malgré la conservation de leur séquence d'ADN).
