- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
L’environnement chromatinien des gènes symbiotiques
Un rôle des régulations épigénétiques et des lncRNAs pour orchestrer l’expression des gènes symbiotiques chez M. truncatula ?
Les avancées pour déchiffrer l’architecture fonctionnelle des génomes eucaryotes ont été récemment facilitées par les progrès technologiques dans les techniques de séquençage, qui ont permis une représentation plus appropriée non seulement des gènes codants, mais aussi des éléments répétés et des ARN non-codants dans les assemblages de séquences génomiques.
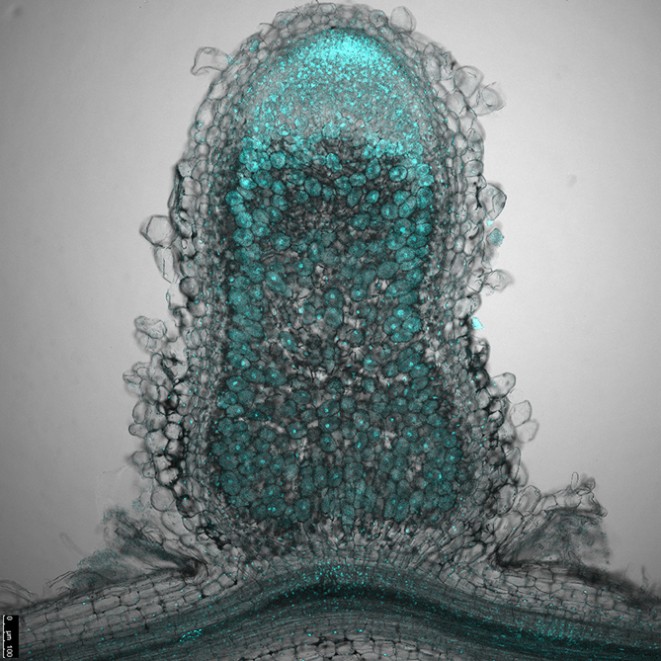
Des équipes des instituts LIPM (Toulouse) et IPS2 (Paris-Saclay) ont utilisé le séquençage PacBio pour réaliser un meilleur assemblage du génome de la plante modèle légumineuse Medicago truncatula (génotype A17), qui a été utilisée notamment pour l’étude des endosymbioses, permettant ainsi d’identifier des réarrangements génomiques entre différents génotypes avec une résolution proche de la base nucléotidique.
L’annotation de ce nouveau génome de M. truncatula a permis pour la première fois une analyse globale des éléments transposables et de leur dynamique. Il a également été possible d’identifier de nouveaux éléments associés au développement des nodosités symbiotiques, en particulier 1037 ARNs non-codants longs (lncRNA) induits dans ces organes.
Il est important de mentionner que nous avons aussi identifié qu'une proportion substantielle (~35% et 38%, respectivement) des gènes induits dans les nodosités ou exprimés dans leur zone de différentiation co-localisent dans des “clusters” génomiques (270 et 211, respectivement), nommés “ilots symbiotiques”. Ces ilots contiennent de nombreux gènes de lncRNA exprimés et présentent à la fois des marques différentielles de méthylation de l’ADN et d’histones.
Les régulations épigénétiques et les lncRNAs semblent ainsi de nouveaux éléments attractifs pour orchestrer l’expression des gènes symbiotiques chez M. truncatula.
Pecrix et al., Nat Plants. 2018 doi: 10.1038/s41477-018-0286-7
Contact : Martin Crespi
