- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Comment la « matière noire » du génome régule-t-elle les gènes distants ?
Un long ARN non-codant affecte la conformation 3D de la chromatine de loci distants par le biais d'hybrides ADN-ARN ou « R-loop »
Au cours des dernières années, une grande partie du transcriptome eucaryote qui ne code pour aucune protéine fonctionnelle, les ARN dits non-codants, sont apparus comme des régulateurs contrôlant l'expression des gènes. Cette « matière noire » du génome eucaryote, bien que non traduite en protéines, peut être transcrite en ARN non-codants fonctionnels participant à un large éventail de mécanismes moléculaires au cours de l'évolution. Au cours de travaux antérieurs, Martin Crespi et ses collègues de l'Institut des Sciences des Plantes de Paris-Saclay (IPS2) ont révélé que le long ARN non-codant APOLO était capable de contrôler l’expression d’un régulateur clé de la signalisation des hormones végétales (le gène PID) en cis par la modulation de l'accessibilité du promoteur du gène PID (Ariel et al., Molecular Cell 2014). Cette année, dans un travail également publié dans Molecular Cell (en ligne le 14 janvier), la même équipe en collaboration avec des collègues de l'Institut d'« Agrobiotechnologie du Littoral » (IAL, Argentine), dans le cadre du LIA CNRS NOCOSYM (Laboratoire International Associé) ont démontré que le long ARN non-codant APOLO peut coordonner l'expression de multiples gènes distants en trans chez Arabidopsis. APOLO reconnaît ces cibles grâce à une complémentarité de séquence et à la formation de duplex d'ADN-ARN appelés « R-loops ».
En utilisant plusieurs techniques à l'échelle du génome, les auteurs ont pu montrer que, lors de la reconnaissance des cibles, APOLO peut moduler la conformation tridimensionnelle de la chromatine des loci distants afin de contrôler finement leur transcription. Cette reconnaissance, dépendante des « R-loops », peut leurrer des régulateurs épigénétiques clés (tels que la LHP1, une protéine impliquée dans les complexes liés à Polycomb) afin de moduler l’accessibilité chromatinienne de ces sites distants. Ainsi, l'activité du génome non-codant façonne dynamiquement l'organisation spatiale de l'information génétique dans le noyau à différents loci.
Cette étude a révélé un nouveau mécanisme de régulation impliquant des ARN non-codants qui pourraient contribuer à la grande diversité des profils d’expression génétique observés dans la cellule eucaryote. Ainsi dans « l’univers génomique », la « matière noire » génétique (les ARN non-codants) pourrait réguler « l’éclat » (le niveau d'expression) des « étoiles » (gènes codants des protéines).
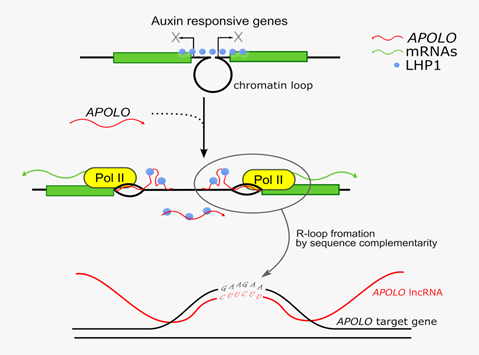
Action en trans du long ARN non-codant APOLO sur un grand nombre gènes répondant à l’auxine.
