- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Projet ANR : PATHOSYM
Comprendre les frontières entre la pathogénicité et la symbiose légumineuse-rhizobia
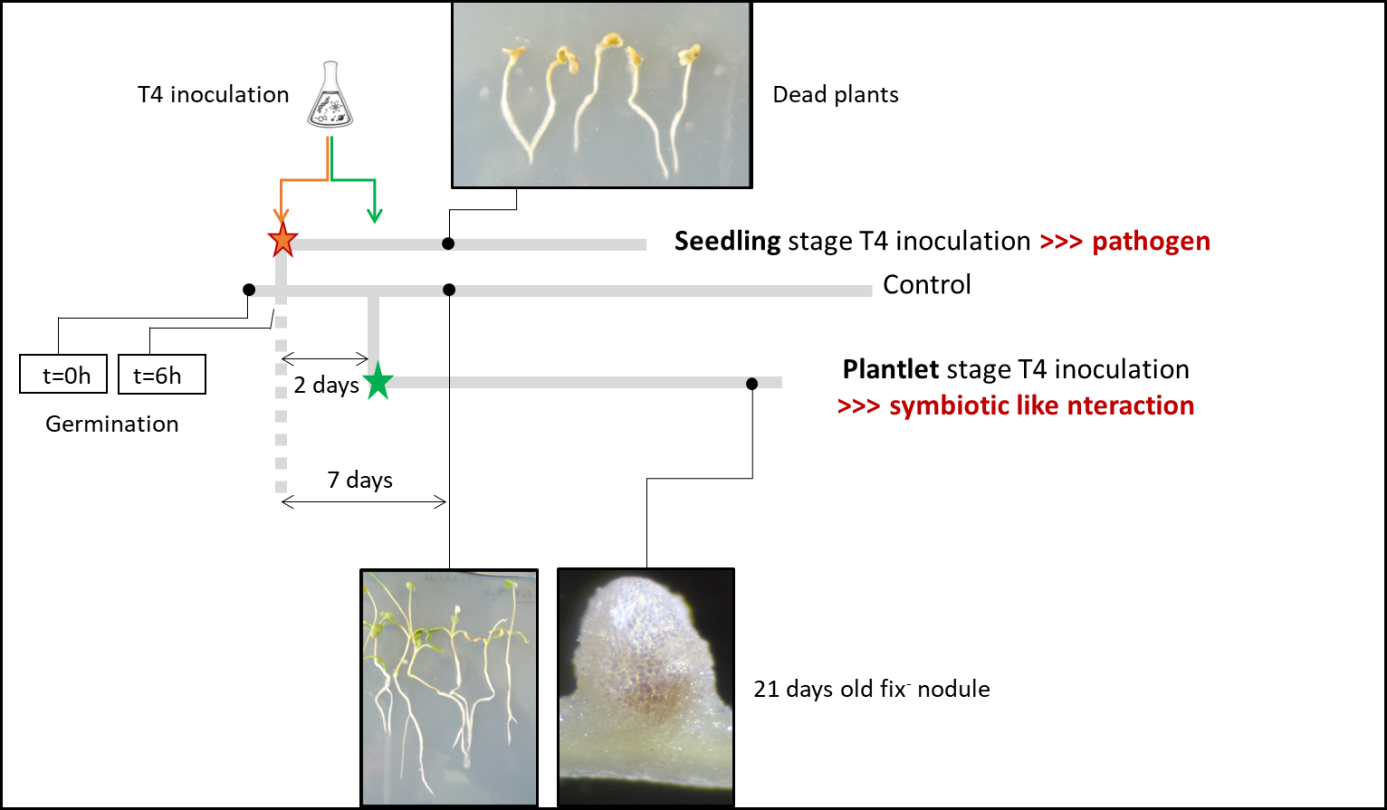
Les légumineuses peuvent établir avec les rhizobia une symbiose fixatrice d’azote palliant le besoin en engrais azoté de la plante. Cette interaction aboutit à la formation de nodosités racinaires où les rhizobia fixent l'azote atmosphérique pour la plante. Les nodosités peuvent aussi être induites par des rhizobia non-fixatrices et/ou être colonisées par des bactéries non-rhizobia. Nous avons isolé de nodules une bactérie atypique (Ensifer adhaerens T4) qui se comporte comme un agent pathogène ou induit des nodosités chez Medicago truncatula.
Le projet ANR PATHOSYM coordonné par Pascal Ratet (Institut des Sciences des Plantes - Paris-Saclay - IPS2, CNRS/INRAE/UPSaclay/UEVE, Gif-sur-Yvette), en collaboration avec B. Gourion et F. Vailleau (LIPME, INRAE, Toulouse) et la startup iMEAN a pour but l’étude de cette interaction originale. Lors de ce projet, nous allons décrire les interactions pathogène et « symbiotique-like » de ce système, puis en intégrant les données génomiques, transcriptomique, protéomique et physiologiques de la souche, construire un modèle du fonctionnement du génome de T4. Nous étudierons aussi la réponse de la plante lors des interactions « symbiotique-like » et pathogène avec la souche T4 ou des dérivés mutants. Le projet PATHOSYM vise donc à décrire les bases moléculaires des interactions T4-M. truncatula pour comprendre les comportements contrastés de T4 et voir comment la plante différencie agents pathogènes et symbiontes.
Mise en ligne 13/01/2022
