- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Un long ARN non codant module le transcriptome par son interaction avec plusieurs facteurs centraux de l’épissage.
L'épissage alternatif (EA) est une source de diversité majeure pour le transcriptome. Les longs ARNs non codants (lncARNs) ont récemment émergé comme des régulateurs de l’EA à travers différents mécanismes moléculaires. Chez Arabidopsis thaliana, les protéines Nuclear Speckle RNA binding (NSRs) qui régulent l’EA interagissent avec le lncARN ALTERNATIVE SPLICING COMPETITOR (ASCO). Dans une étude parue dans EMBO reports (Rigo et al., 2020), l’équipe REGARN de l’IPS2 a analysé l’effet de la suppression et de la surexpression d’ASCO à l’échelle globale du transcriptome, démontrant qu’un nombre conséquent de gènes liés aux réponses de défense des plantes présente une expression dérégulée ou un épissage altéré. De plus, des plantes présentant une réduction d’expression d’ASCO sont plus sensibles à la flagelline, une molécule mimant une attaque par un pathogène. Grâce à l’utilisation d’une méthode de purification de ribonucléoprotéines basée sur l’usage d’oligonucléotides biotinylés, l’équipe a pu identifier de nouvelles protéines associées à ASCO, mettant en lumière l’interaction entre cet ARN et une protéine clé fortement conservée de la machinerie d’épissage : Pre-mRNA PROCESSING 8a (PRP8a). De plus, la suraccumulation d’ASCO altère la reconnaissance des transcrits cibles de PRP8a, suggérant qu’ASCO se lie directement au cœur de la machinerie de l’épissage. En effet, il a également été démontré qu’ASCO est capable d’interagir avec un autre élément très conservé de la machinerie d’épissage, la protéine Small nuclear ribonucleoprotein Sm D1b (SmD1b). Ce résultat indique qu’ASCO est capable d’interagir avec, en plus des NSRs, des facteurs centraux de la régulation de l’épissage, de la même manière que les petits ARNs nucléaires U1 à U6. Ainsi, les lncARNs pourraient faire partie d’un large et dynamique réseau de régulation impliquant des composants très conservés de la machinerie d’épissage. L’une des questions majeure qui demeure est de comprendre comment ces lncARNs très divergents au niveau de leur séquence nucléotidique peuvent interagir avec le cœur très conservé de la machinerie de l’épissage. Ceci laisse entrevoir de nouveaux mécanismes par lesquels les lncARNs pourraient moduler la reprogrammation du transcriptome chez les Eucaryotes.
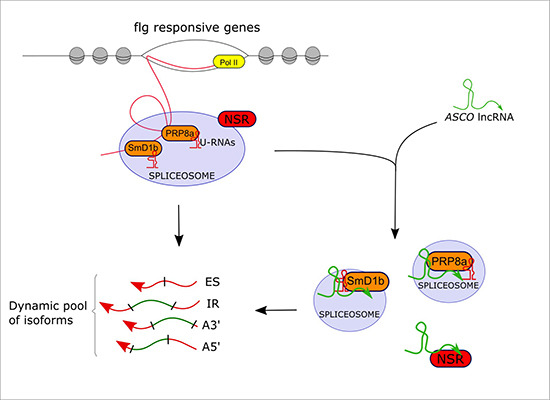
Modèle schématique représentant le rôle du lncARN ASCO dans la modulation de l’épissage alternatif de transcrits spécifiques.
Le lncARN ASCO interagit avec les facteurs d’épissages fortement conservés PRP8a et SmD1b afin de moduler l’épissage alternatif de transcrits liés à la réponse à la flagelline.
