- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Un peptide CLE inattendu régule la nodulation symbiotique des plantes légumineuses
Un test statistique amélioré basé sur le déséquilibre de liaison et utilisé pour des scans de sélection épistatique à l'échelle du génome a permis de prédire une interaction génétique entre un nouveau peptide de signalisation CLE et le récepteur SUNN
La recherche de signatures de sélection à l'aide de données de « polymorphisme nucléotidique unique » (SNP) s'est avérée efficace pour découvrir des gènes impliqués dans des fonctions moléculaires conservées et / ou adaptatives, mais aucune méthode statistique n'a été conçue pour identifier les allèles en interaction comme cibles de processus sélectifs. Dans une étude collaborative entre l’équipe Frugier de l'IPS2 et M. Bonhomme du LRSV (Toulouse, France), publiée dans Heredity, nous proposons un test statistique visant à détecter la sélection épistatique qui se base sur une mesure de déséquilibre de liaison (LD) tenant compte de la structure de la population et des relations hétérogènes entre les individus. Les statistiques basées sur les SNPs individuels (Trv) et sur une fenêtre de SNPs (TcorPC1v) correspondent toutes deux à une distribution de Student, ce qui permet de tester la significativité des coefficients de corrélation. L'utilisation de ce test statistique amélioré a permis d'utiliser les données SNP de la légumineuse symbiotique Medicago truncatula pour découvrir une coadaptation génétique jusque-là inconnue entre le récepteur MtSUNN (« Super Numeric Nodule ») et le peptide de signalisation MtCLE02 (« CLAVATA3-Like 02»). Des preuves expérimentales ont permis de démontrer à titre de preuve de concept que cette signature de coadaptation était associée à un lien génétique entre ces deux gènes, puisque les peptides de signalisation MtCLE02 régulent négativement la nodulation symbiotique de manière dépendante du récepteur MtSUNN. Ce travail collaboratif a été rendu possible grâce au projet ANR «PSYCHE» coordonné par F. Frugier à IPS2.
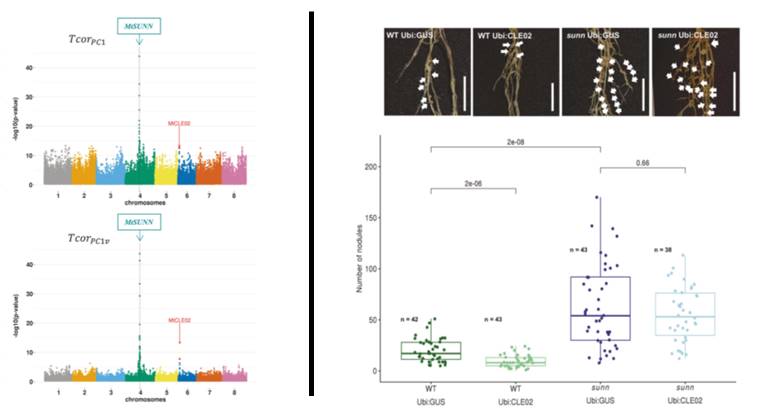
Figure. De l'identification d'une signature de coadaptation entre deux gènes de Medicago truncatula à la preuve expérimentale de leur interaction génétique.
A gauche, la distribution LD calculée à partir de la statistique TcorPC1 (au-dessus) et de
la statistique TcorPC1v (en dessous) entre le gène appât MtSUNN (encadré) et tous les gènes du génome de M. truncatula. L'axe des abscisses correspond aux positions des gènes couvrant les huit chromosomes, chaque point correspondant à un gène et le gène MtCLE02 étant indiqué par une flèche. L'axe des y montre le -log10 (valeur p) du test du coefficient de corrélation.
A droite, nombre de nodules symbiotiques formés sur des racines témoins (Ubi: GUS) ou des racines surexprimant le peptide de signalisation CLE02 (Ubi: CLE02), dans des plantes sauvages (« WT » ; deux images et barres de gauche) et dans un mutant du récepteur SUNN (« sunn » ; deux images et barres de droite). Ces résultats démontrent que l'effet négatif de CLE02 sur le nombre de nodules nécessite l'activité du récepteur SUNN.
