- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
HDR de Véronique BRUNAUD
Véronique Brunaud a soutenu son HDR le 24 novembre 2022
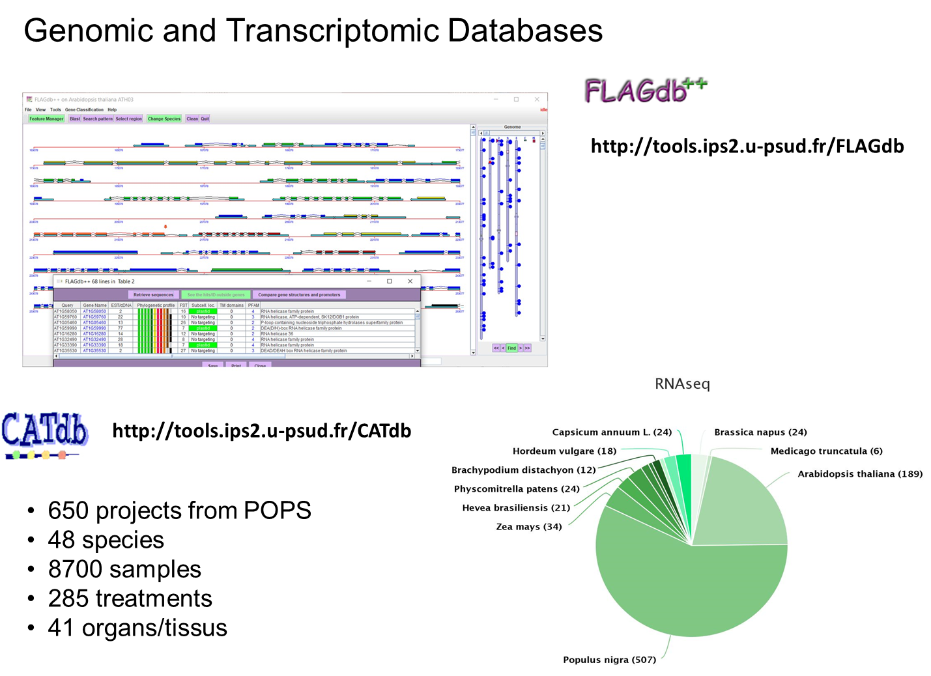
Gestion et intégration de données génomiques pour l’étude de la transcription chez les plantes
Mes activités en tant qu’ingénieure de recherche en bioinformatique s’inscrivent essentiellement sur deux axes : (1) la création et le développement de systèmes d’information pour traiter les données venant de plateformes ou de projets scientifiques centrés sur la génomique et la transcriptomique végétales (bases de données FLAGdb++ et CATdb) ; (2) la régulation de la transcription chez les végétaux et des mécanismes d’interactions entre les facteurs de transcription et leurs gènes cibles. La méthode « PLMdetect », mise au point dans l’équipe GNet de l’IPS2, permet de détecter des motifs d’ADN cis-régulateurs qui servent d’ancrage aux facteurs de transcription. Cette méthode est maintenant utilisée dans plusieurs projets de recherche dont celui que j’anime sur la détection de facteurs de transcription co-actifs lors de stress environnementaux.