- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Les règles de régulation transcriptionnelle sont sélectionnées pour une complexité minimale
La minimisation de la complexité détermine les règles de régulation transcriptionnelle dans les modèles booléens de systèmes vivants
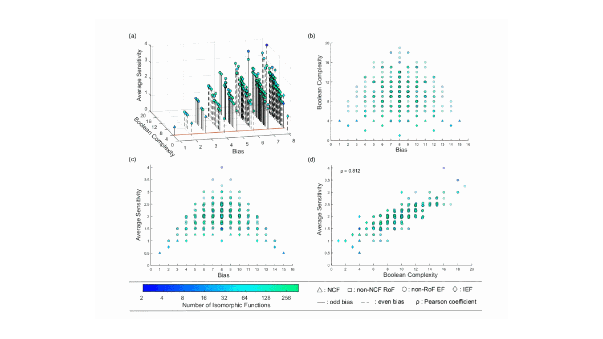
Les « décisions cellulaires » dépendent de l’action coordonnée de multiples composantes moléculaires. Leur modélisation peut être traitée par une approche de type réseaux booléens où des arêtes orientées représentent les dépendances et où une règle logique spécifie comment l’état d’un gène est déterminé par l’état de ses régulateurs. Des données sur les interactions entre les composantes de tels réseaux permettent la construction de modèles de régulation booléens. Bien que la structure de ces réseaux ait été largement explorée, il a peu d’études sur la caractérisation des règles logiques associées.
Dans une étude publiée dans PNAS Nexus, un chercheur de l’IPS2 (équipe REGARN) et une équipe au IMSc, Chennai (Inde), ont compilé 2687 règles logiques publiées pour 88 réseaux de régulation booléens. Ils trouvent que ces règles satisfont généralement les propriétés attendues telles que monotonicité et canalisation. Les auteurs trouvent aussi que deux types de règles sont très sur-représentées : les fonctions « read-once » et les fonctions canalisantes imbriquées. Or ces deux types ont une complexité minimale si on se réfère à la complexité booléenne ou à la sensibilité moyenne. Cette analyse indique donc que les règles les plus simples sont utilisées prioritairement dans les décisions cellulaires.
19/05/2022
