- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Un modèle quantitatif de la répartition des crossovers
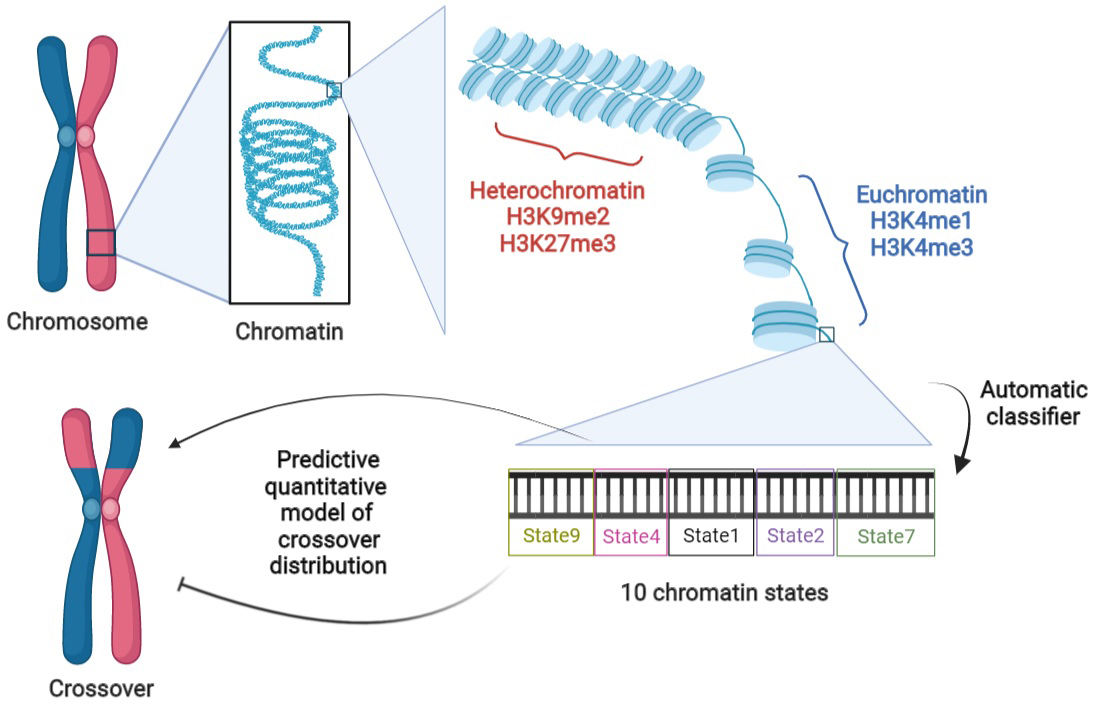
Lors de la méiose, un processus de formation de cassures puis de réparation de l’ADN conduit à la formation de crossovers entre chromosomes homologues. Ces chromosomes recombinés sont transmis à la descendance et permettent un brassage génétique sur lequel agit la sélection naturelle ou artificielle. Cependant, les facteurs controlant le nombre et le positionnement des crossovers restent incompris. Arabidopsis thaliana est la plante avec les jeux de données les plus informatifs pour les crossovers, avec des données génomiques et épigénétiques publiques abondantes, ce qui permet d’étudier les caractéristiques des crossovers et d’en tirer de nouvelles connaissances par modélisation.
Dans une étude publiée dans Quantitative Plant Biology, des chercheurs de l’IPS2 (équipe REGARN) et de GQE-Le Moulon ont construit un modèle quantitatif de la répartition des crossovers le long des chromosomes tenant compte de l’état de la chromatine et du contenu génomique. Les régions intergéniques avec un statut épigénétique euchromatique sont apparues comme étant enrichies en crossovers par rapport aux régions géniques ou hétérochromatiques. De plus, la densité des crossovers par Mb est réduite dans les régions intergéniques de faible taille (plus petite que 1,5 kb) et dans les régions où les séquences des chromosomes homologues sont identiques. Au final, les auteurs ont construit un modèle quantitatif et prédictif reproduisant la plupart les variations des profils de recombinaison dans A. thaliana.
Mise en ligne le 28/03/2022
