- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
La plateforme de transcriptomique POPS : des analyses ARN à façon
Focus sur la plateforme POPS
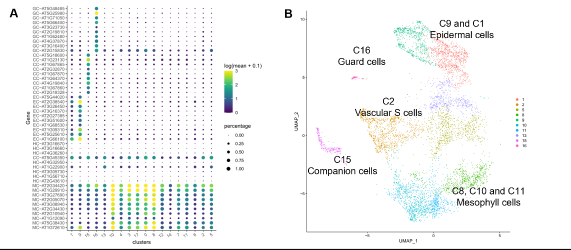
La Plateforme de Transcriptomique des Plantes de Paris-Saclay (POPS) propose aux laboratoires académiques ou privés des outils d'analyse du transcriptome par séquençage à haut débit de l'ARN ou « RNA-seq ». Nous sommes spécialisés dans l'analyse des plantes modèles ou de culture. Le service proposé inclut la génération des données de « RNA-seq », les analyses bio-informatiques et statistiques, ainsi que des conseils pour l'interprétation des résultats. Nous vous accompagnons donc depuis le design expérimental jusqu'à l'interprétation des données. La plateforme est certifiée pour l'ensemble de ses activités selon les exigences de la norme ISO 9001 depuis 2012. Afin d'offrir à chaque collaborateur l'analyse la plus adaptée à ses questions biologiques, nous disposons des équipements et des outils d'analyses bio-informatiques et statistiques permettant la construction de transcriptome de novo, l'analyse d'accumulation des ARNs poly-adénylés ou non, des petits ARNs (miRNAs, siRNAs, ribo-seq...), et des ARN poly-adénylés à partir de très petites quantités de départ (jusqu'à 50pg). Pour les analyses d'expression tissu-spécifiques, nous offrons également des analyses de transcriptome après micro-dissection laser ou en cellule unique grâce à la technologie Chromium (10x Genomics). En collaboration avec l’équipe STRESS de l’IPS2, nous avons par exemple étudié le comportement individuel des cellules d’une feuille d’Arabidopsis thaliana lors d’une infection par la bactérie phytopathogène Pseudomonas syringae. En utilisant la technologie 10X Genomics, nous avons pu mettre en évidence l’hétérogénéité des réponses végétales au sein de différentes populations cellulaires, permettant ainsi de définir un nouvel et original champ de bataille entre plante et pathogène (Delannoy et al., bioRxiv 2023).
Contact : pops.ips2 @ universite-paris-saclay.fr
Plug In Labs Université Paris-Saclay : cliquer ici
09/05/2023
