- Présentation
- Recherche
- DGG : Département Génomique et Génétique du Développement
- REGARN : Les ARN non-codants, des acteurs de la plasticité développementale de la racine
- ChromD : Dynamique des chromosomes
- SILEG : Voies de signalisation contrôlant le développement du système racinaire des légumineuses
- FLOCAD : Développement floral et déterminisme du sexe
- Qlab : Equipe Génomique et épigenomique quantitative des plantes
- DPHYS : Département Physiologie et Signalisation
- PMIN : Département Interactions Plantes Micro-organismes et Réseaux
- DGG : Département Génomique et Génétique du Développement
- Enseignement
- Plateformes
- Bases de données
Transcriptomique à grande échelle et à moindre coût
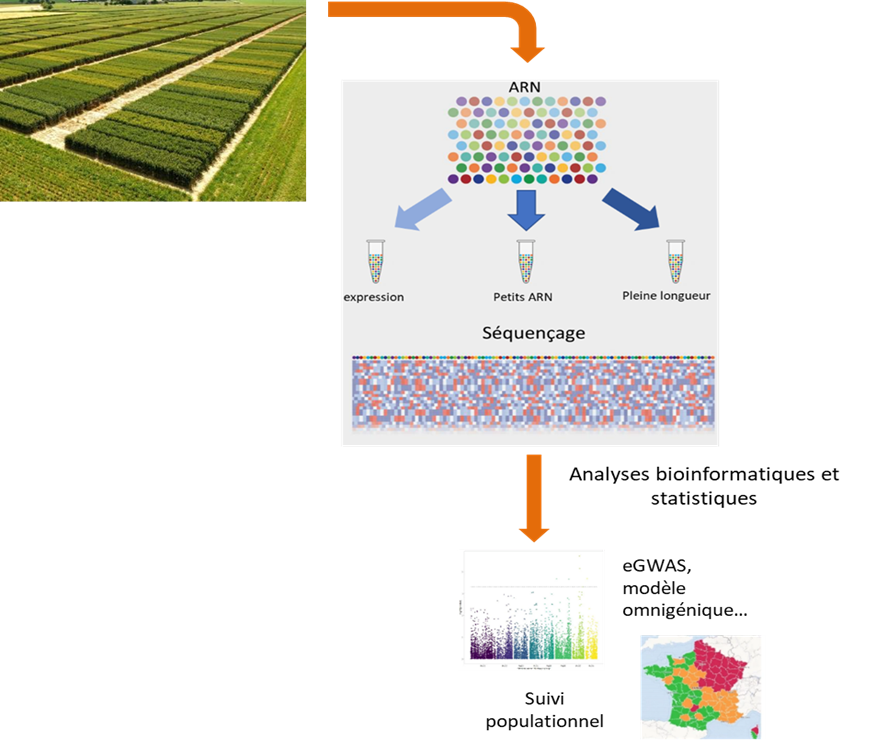
L’analyse de transcriptome est largement utilisée dans les projets de recherche. En offrant une vision exhaustive de l’activité des gènes d’un organisme, la transcriptomique permet d’explorer et d’identifier les mécanismes expliquant les phénotypes observés. Cette approche peut s’appliquer à n’importe quel échantillon (tissu ou espèce), cependant son utilisation reste souvent cantonnée à des questions relevant de « designs » expérimentaux relativement simples. Il y a probablement deux raisons à cela. La première est la masse très importante de données générées qui nécessite une analyse complexe et experte. D’aucuns pensent que l’intelligence artificielle et les réseaux de neurones pourront lever ce verrou, mais ces approches nécessitent une quantité énorme de mesures pour espérer identifier les structures explicatives et prédictives parmi les dizaines de milliers de transcrits analysés dans chaque échantillon. De même, les problématiques de l’agroécologie exigent des analyses faites au champ dans des conditions bien plus complexes (multiples espèces, multiples facteurs) et hétérogènes qu’au laboratoire. Cela nécessite là encore l’analyse d’un très grand nombre d’échantillons. Afin d’apporter la puissance et la sensibilité de la transcriptomique à ces problématiques majeures, il faut donc la rendre accessible au plus grand nombre et en réduire drastiquement les coûts. La plateforme transcriptomique POPS offre à présent un service de « screening » de transcriptomes végétaux à très haut débit (plusieurs centaines d’échantillons par projet) et à prix très réduit, grâce à la technologie BRB-Seq (Bulk RNA barcoding and sequencing) qui permet la réduction des volumes réactionnels ainsi que l’automatisation de toutes les étapes (extraction d’ARN, fabrication des banques, dosages et multiplexage). Nos équipements nous permettent également de réduire les volumes réactionnels.
03/11/2025
